これはWindows10 ユーザー向けに、RNA-SeqのFASTQファイルをSubio Platformにインポートするための準備について説明したものです。もしMacをお使いでしたらmacOS版の説明をご覧ください。
Windowsコンピューターで、Linux用のプログラムを実行することになりますので、Windows Subsystem for Linux を有効化することと、Ubuntu (18.04 or 20.04) をインストールすることが必要です。WSLはWindows 10から使える機能です。Windows 11の場合、WSLのバージョンでWSL1またはWSL2を選択できますが、どちらでも動作します。
下のムービーは、Windows 10でのセットアップ手順をご覧いただけます。Windows 11をお使いでしたら、Ubuntuのインストールは参考ページをご覧ください。LinuxのGUI packageは必要ありません。
現在、fastpのインストール手順がムービーでご覧いただけるものとは変わっています。anacondaアーカイブからfastpの実行ファイルを取得するのが簡単です。詳細は、このページの最後の段落をご覧ください。
Subio Platformは、下記のツールを使ってRNA-SeqのFASTQファイルを処理します。
手順:
- WSLを有効化する。
- マイクロソフトのアプリストアから、Ubuntuを入手する。
- Ubuntuを起動し、初期化する。Linuxシステム用のユーザー名とパスワードを設定する必要があります。
- fastp、HISAT2、StringTieについて、それぞれのLinux版バイナリーファイルをダウンロードする。
- Subio Platformの設定画面で、これらツールのバイナリー(executable)ファイルのパスを設定する。
- 目的の生物種のHISAT2インデックスファイル をダウンロードする。
- 目的の生物種の、HISAT2インデックスと同じゲノムバージョンのGTFファイルをダウンロードする。
- Subio Platformの設定画面で、HISATインデックスと、GTFファイルのパスを設定する。
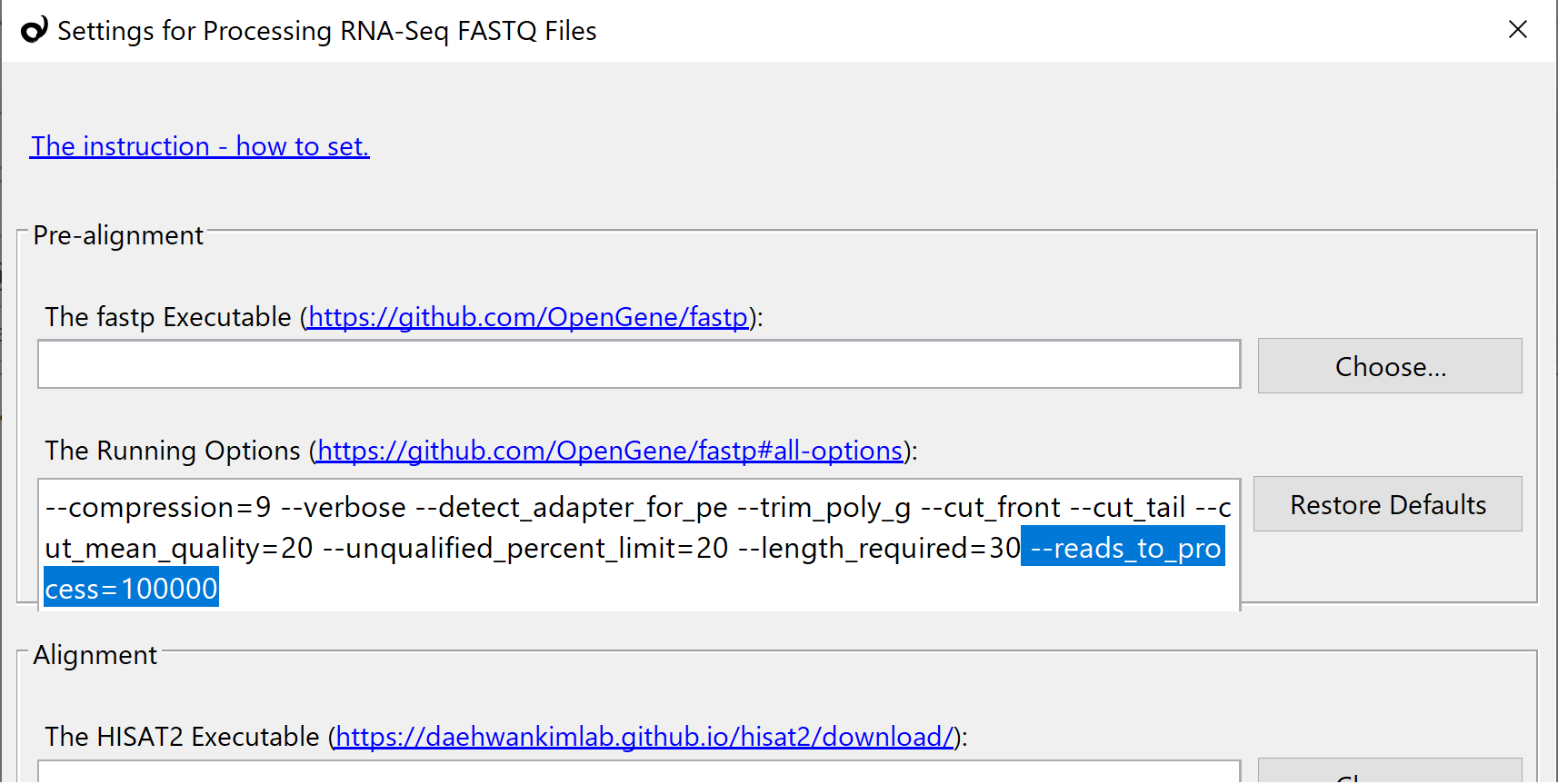
FASTQファイルを処理するのにはとても長い時間がかかります。そこで、パイプラインの設定がうまくいったかどうか短い時間で試すために、下記のオプションをfastpのセクションに追加してください。こうすることで、処理するリードの数を制限することができ、FASTQ処理を実行しても数分で完了します。うまくいくことを確認したら、このオプションを削除して、すべてのリードを処理してください。
--reads_to_process=100000
上記で紹介したツールは、それぞれ下記のバージョンで正しく実行できることを確かめています。これと異なるバージョンの場合、エラーが起きる可能性があります。
- fastp 0.22.0
- HISAT2 2.1.0
- StringTie 2.1.1
ちなみに、HISAT2 2.2.0を使う場合はちょっとだけ注意が必要で、indexやGTFのパスからすべてのスペースを除く必要があります。
そしてfastpは0.22.0をお勧めします。他のバージョンだとSubio Platformのパイプラインでエラーが発生するかもしれません。インストールしたバージョンのfastpで問題が発生する場合は、Anacondaのfastpアーカイブから、linux-64/fastp-0.22.0-h2e03b76_0.tar.bz2 をダウンロードしてfastpファイルを置き換えてください。