遺伝子の発現量を測定する技術は、少なくとも2004年までには現在の水準に達していますので、ちゃんとしたスキルを持つ実験者により測定されたデータであれば、信頼できると言えます。一方で、マイクロRNAの発現量の測定は今なお難しいです。miRNAの発現データが、遺伝子発現データと同等に信頼できるものではないということは知っておいたほうが良いでしょう。ここでは、hepatocellular carcinoma (HCC) の複数のデータセットに跨って、遺伝子およびmiRNA発現データを見比べてみます。
遺伝子発現データセットの例
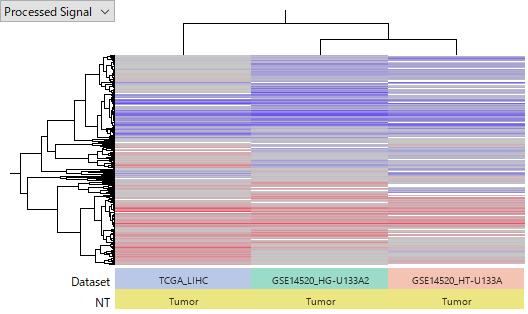
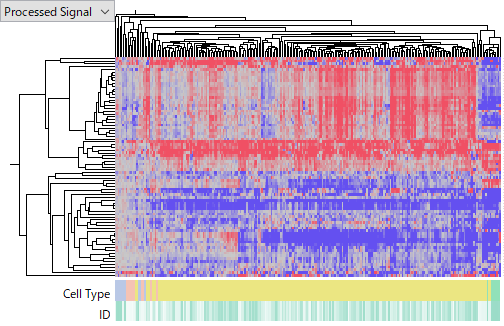
下のヒートマップは、TCGA-LIHC と GSE14520 という二つのデータセットを並べて見比べるためのものです。TCGAの遺伝子発現データは、RNA-Seqによりとられています。一方 GSE14520 はAffymetrix HG-U133A 2.0 Array と HT_HG-U133A Array という2種類のGeneChipを使って測定されたものですので、合計3つのデータセットとして扱っています。生データから処理し直して、Tumor サンプルにおける Normalサンプルに対する Log2 Ratio に変換して解析しました。赤い色はTumorで発現が亢進していることを、青色は減退していることを表します。
異なる研究者ならびに異なる手法により測定であるにもかかわらず、この3つのデータセットにおいて発現の上昇と下降がおおよそ一致しています。即ちこれらのデータの信頼性は高いといえるでしょう。
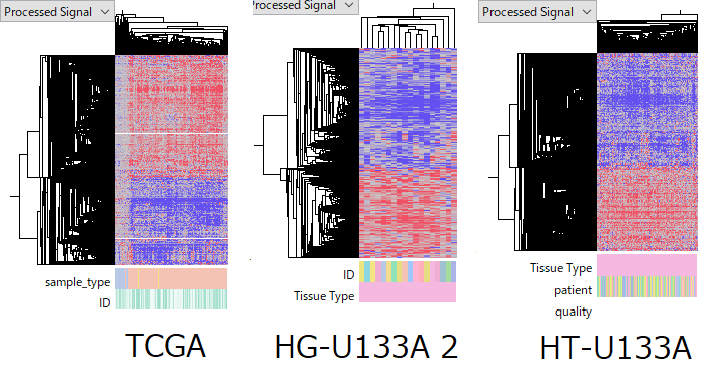
各データセット内の一貫性
TCGA-LIHC RNA-Seq データセットは、50 の Normal と 370 の Tumor サンプルで構成されています。GSE14520 HG-U133A 2.0 Array データセットは、18 の Normal-Tumor ペアを含んでおり、HT-U133A データセットでは 214 ものペアを含んでいます。いくつか品質に疑問のあるサンプルが含まれるものの、全体としては質の高いデータセットと言えます。
それぞれのデータセットを見てみても、Tumor サンプルの発現プロファイルがだいたい似通っているようにみえます。これにより、これらの発現データが本来の生物学的状態を反映しているという主張に合理性をもたらします。
microRNA 発現データセットの例
次に、miRNA の発現データセットを見てみましょう。遺伝子発現データセットのときと違い、各データセットの結果がほとんど一致していません。miRNA発現データの解釈がそう簡単にはいかないことがうかがい知れます。

各データセット内の一貫性(または不一致)
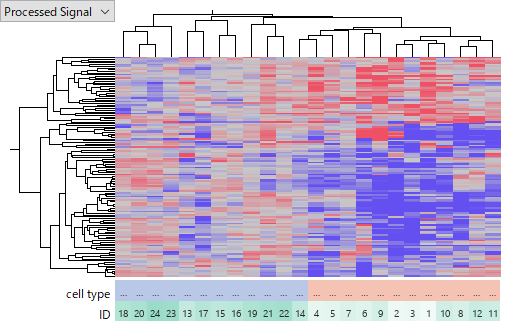
これらのデータセットで、一番大きなのはTCGA-LIHC miRNA-Seq のデータです。ヒートマップでは Tumor サンプルにおける miRNA の発現プロファイルがおおよそ一致しており、この実験がうまくいっているように見えます。
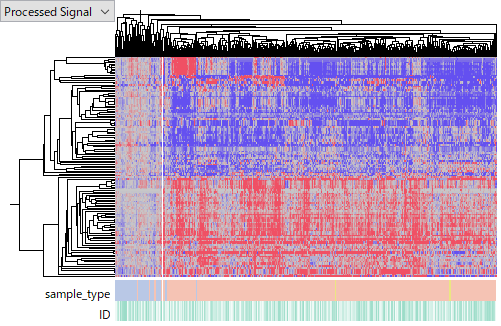
GSE110217 は、Agilent のHuman miRNA v16 microarray を使って測定されたものです。このデータセットはデータの品質に問題がありそうで、繰り返しサンプルの後半 (5 - 8) で、明らかに前半 (1 - 4) よりもシグナルが低いです。実験者のスキルが十分でなかったのではないかと考え、後半のサンプルを解析から除外しました。GSE110217は、Normal、non-HCC、HCCの3つのグループを含んでいますが、クラスタリングの結果を見るとHCCのサンプル群が右側にクラスターを形成しており、生物学的コンテキストから合理的に見えます。つまり、これも実験はうまくいっているようです。
しかし、上のデータセットを比較したヒートマップを見ると、発現上昇(あるいは下降)している miRNA がTCGAの結果をまったく一致していません。異なるプラットフォーム由来のデータセットを、miRNAの名前で名寄せして統合する際に幾分不整合があり、これにより一部でうまく比較できない可能性があるとはいえ、上の結果はあまりにも一致しなさすぎます。
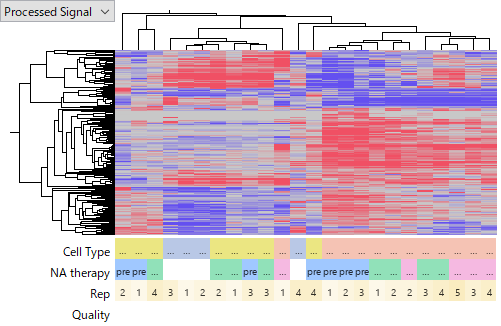
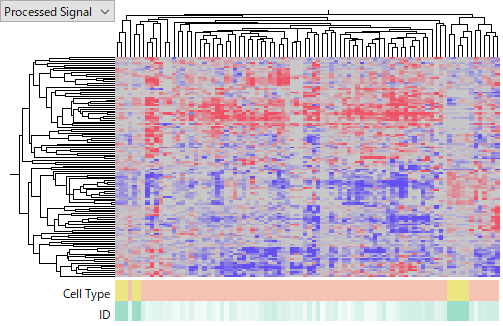
この後のヒートマップは GSE74618 (Affymetrix miRNA v2 Array)、
GSE115016(Affymetrix miRNA v4 Array)、
GSE10694(CapitalBio Mammalian miRNA Array)、
そして、GSE28854(Milteny Biotec miRXplore miRNA Microarray)の各データセットのものです。
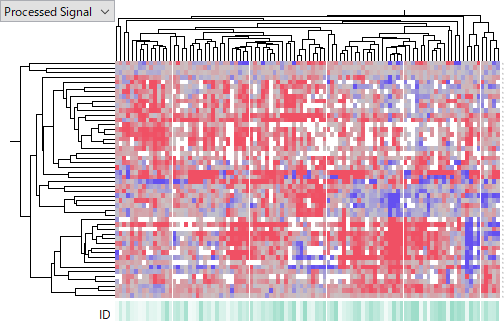
より後のヒートマップほど、HCCサンプル間での違いが大きい、あるいはノイズの大きいデータに見えます。しかし、ここではどの測定方法が良いかを論じたいわけではありません。測定システムの良し悪しはもちろん、実験者のスキルのばらつきや、その他の見えていない要因が影響している可能性もあるからです。ここで言えるのは、miRNAの発現データは遺伝子発現データほど信頼できないということです。上の例でも、結局HCCで発現が上昇している(あるいは下降している)miRNAはどれか、という問いに答えるのがいかに難しいかわかるでしょう。
この難しさは、miRNAの次の性質に由来します。
- 遺伝子に比べて、miRNAの塩基配列が短く、しかも近似度が高い。
つまり、それぞれのmiRNAについて発現量を正確に安定的に測定するのが非常に難しいということです。 - 遺伝子に比べて、miRNAの数が圧倒的に少ない。
つまり、測定データを正規化することが困難で、どのような処理をしても必ず疑問が残るということです。
以上より、miRNAの発現量の網羅的測定は、簡単には解決できない難題を抱えている発展途上の技術といえるでしょう。miRNAのデータセットを使う時、または論文で報じられた発現上昇している(あるいは下降)しているmiRNAのリストを使う際には、十分な注意が必要です。
もし miRNA の実験を計画しているのでしたら、遺伝子発現の実験以上に高いスキルが必要だということを忘れてはいけません。Subio にご相談されたい場合は Contact us よりご連絡ください。
データをダウンロードして、Subio Platformで詳しく見てみよう
お手元の Subio Platform にデータをインポートして詳しく見てみたい方は、SOA ファイル をダウンロードしてください。SOAは、SSA ファイル を束ねたようなもので、簡単にSubio Platformにデータを展開できます。
操作:
Platform メニューからImport Archive... を開いて、ダウンロードしたSOAファイルを選択してください。インポートが完了すると自動的にSubio Platformが閉じますので、ソフトウェアを再起動してください。