EnsemblのFTPでは、最新版のGTFがcurrentという名前で提供されています。そして、よく見るとたくさんのバージョンがあって、どれでもダウンロード可能です。何かのツールの操作方法を学びながらやっていると、特定のバージョン番号を記述していたりしますが、それが最新版でないときもあります。どちらのバージョンを選択するのがいいのでしょうか?
これについては、どのバージョンでもいいと思います。どのバージョンを使うと本当の遺伝子発現状態に近づけることができるのかなんて、誰にも分かりません。そして、どのバージョンかは問題の核心ではありません。
GTFのバージョン84と99を使って算出された発現量(TPMとcounts)を比べた、下の散布図を見てみてください。ほとんどの遺伝子の発現量はほとんど同じですが、一部の遺伝子については極端に異なる発現量が算出されていることが分かります。
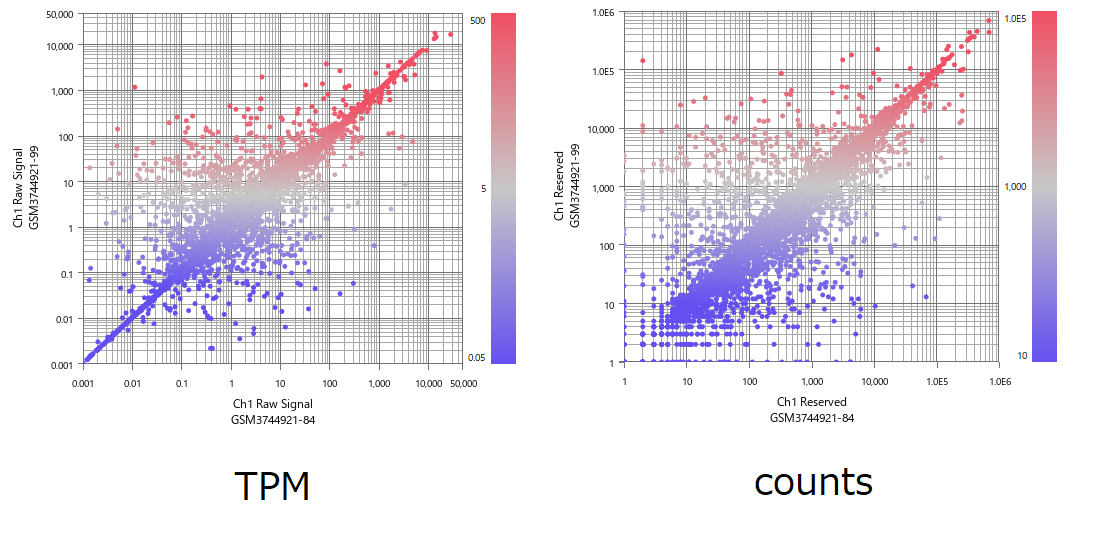
このように、発現量の推定値は、アルゴリズム(そのバージョンと実行オプション)やマッピング対象のリファレンスゲノム、そしてGTFの違いに敏感に反応します。ここで、複数の研究グループから出された発現データをまとめて解析する場面を考えてみてください。これらのデータセットが、完全に同じパイプライン設定で計算されているという仮定は現実的ではありませんよね。
従って、複数のデータセットをまとめて解析する場合、すべてのFASTQファイルを集めて、まとめて処理しなおす必要があるということが分かります。既存のTPMやFPKM、countsなどの数値を混ぜて比較してはいけないのです。
発現が上昇、あるいは下降しているように見える遺伝子でも、それはパイプラインの影響を大きく受けた結果に過ぎません。遺伝子個別のレベルでは、あまり信用できないと考えたほうが良いでしょう。
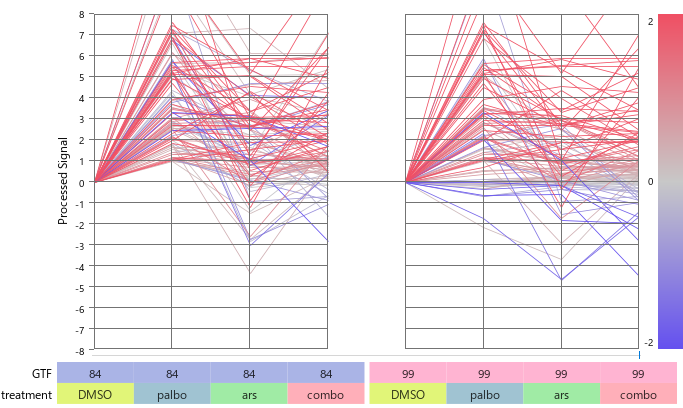
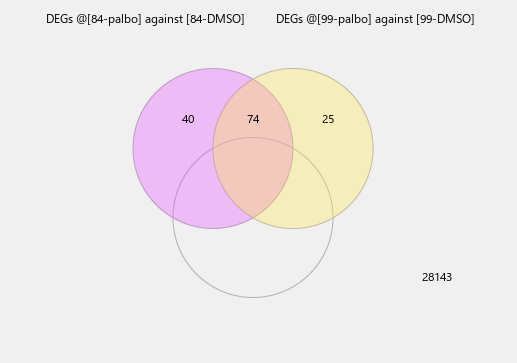
たとえば、GTFのバージョン84と99を使って算出された発現量を解析して得られた、二つの発現上昇遺伝子リストを比べてみると、関集合に入る遺伝子もありますが、ほとんと同じというわけにはいかないでしょう。
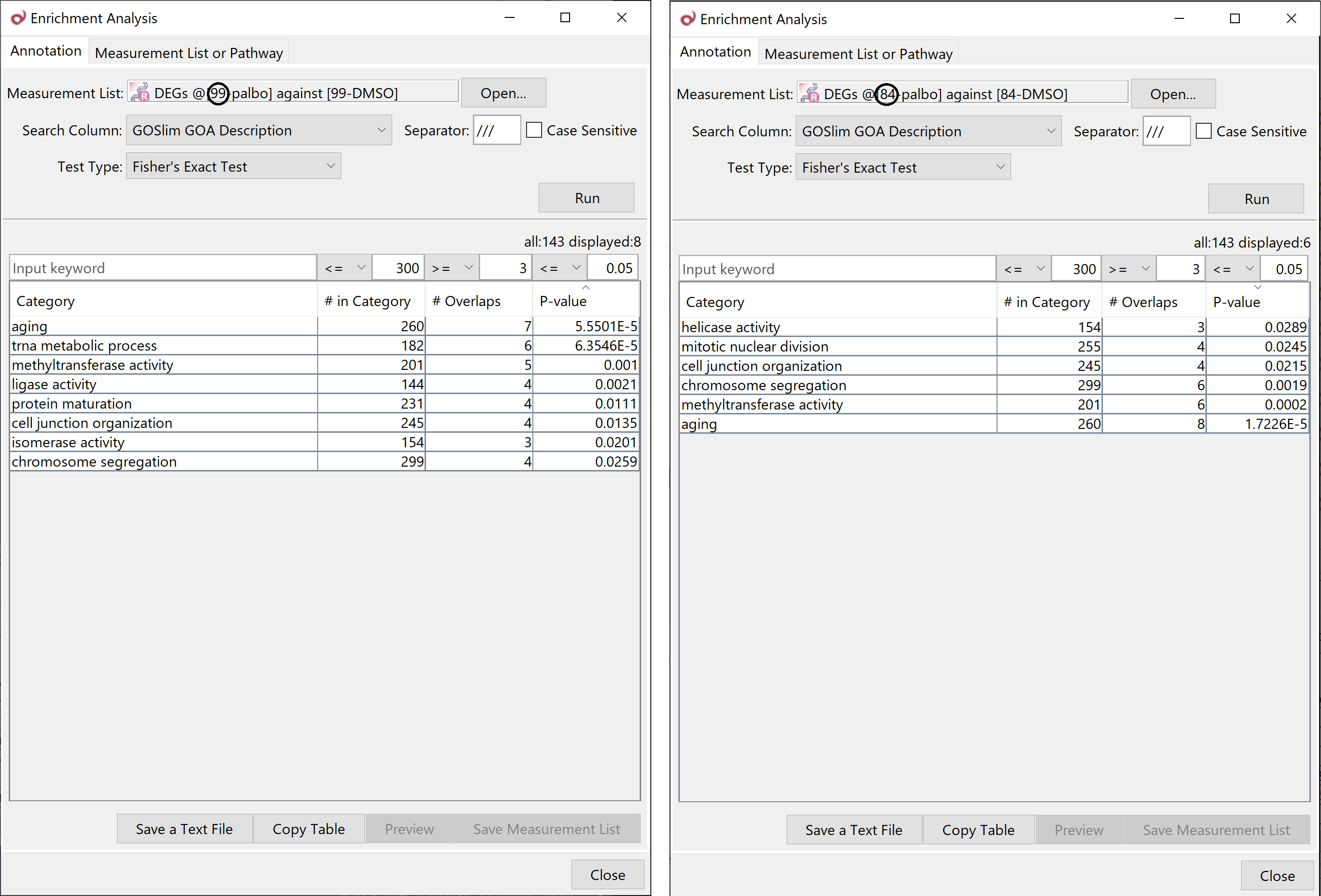
しかしそれでも、この二つの遺伝子リストをエンリッチメント解析にかけた結果はよく似ており、そこから導出される生物学的結論にはあまり違いがない、ということがオミクスの性質から期待できるのです。
このデータのSSAファイルをインポートして、GTFのバージョンの違いによる差をご自身の目でご確認ください。
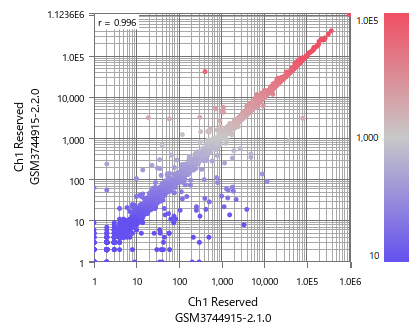
HISAT2のバージョン2.1.0 と 2.2.0で生成されたデータも比較してみました。GTFより差が小さいとはいえ、異なるバージョンを使ったデータを混ぜるのも避けたほうが良いことが分かります。SSAファイルをインポートしてご確認ください。