Ch1RawSignal は Subio Platform にインポートした生の値です。2色法の実験データを取り込んでいたら、Ch2RawSignal もあります。
Processed Signals は、Setup Series タブの Normalization で定義されたプロセスによって、Ch1RawSignal から作られた値です。
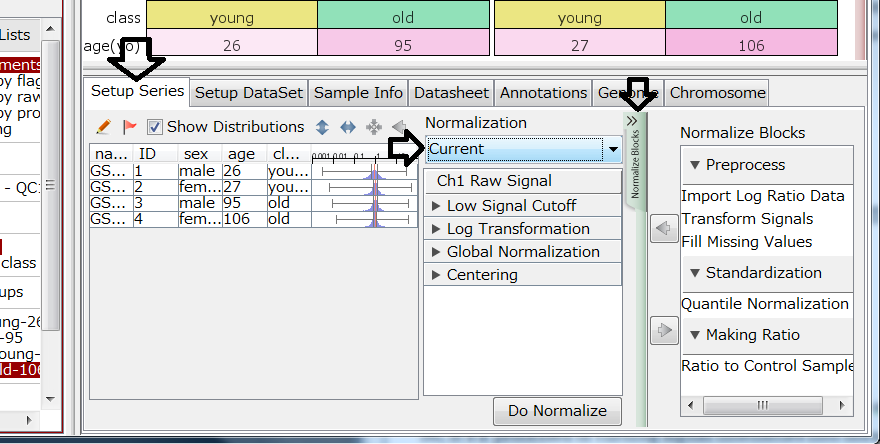
Normalization の編集パネルでは、最上部に必ずCh1RawSignalがあります。ブロックは右の Normalize Blocks という緑色の引き出しタブから追加することができます。ブロックがどのような影響をもたらしたかは、右側のヒストグラム+ボックスプロットでリアルタイムに表されます。そして、最後のブロックを選択しているときに表示されている値が、Processed Signalと同じです。ただし、Do Normalize ボタンを押さない限り、現在の Processed Signal は上書き更新されません。
ですから、安心してノーマライズブロックを追加・削除、順序変更、オプション設定の変更尾などを試行錯誤してみることができます。もしよくわからないことになってしまったら、上のプルダウンメニューから Current を選ぶと、現在適用されている状態に戻すことができます。これでよいという状態になったら、Do Normalize ボタンを押して Processed Signal を更新します。
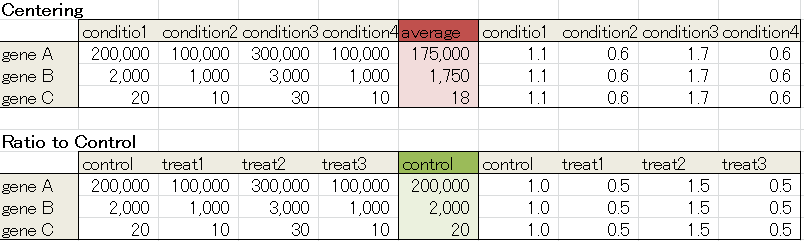
Processed Signalは、一般的には、「正規化されたデータの、対数比」です。centering ブロックが適用されていれば、比の分母はその遺伝子の発現量の平均値です。Ratio to Control Samples ブロックが適用されていれば、分母はそのブロックのオプションで設定したコントロールサンプルの発現量です。
一般的に、発現解析はシグナル値(Ch1RawSignal)ではなく、対数比(ProcessedSignal)のデータに対して行います。
なぜシグナル値を比に変換するのでしょうか?
それは、オミクスデータは非常に複雑なので、シグナルを比に置き換えることにより、発現量という情報を無視して、発現がいつどれくらい変化したかという変動量に単純化できるからです。
具体的に表の数値を見てみてください。Centering ブロックを使おうが、Ratio to Control を使おうが、比にすると全く同じ値になっていることがわかります。ブロックの違いは、分母となる値です。centeringでは、平均値を使っているので、比の値が1より大きくても小さくても大した意味はありません。しかし、Ratio to Control Samples を使っているときは、1より大き(小さ)ければコントロールよりも増えて(減って)いることを表します。
しかし、比に置き換えることはデータを単純化する方法にすぎません。単純化した状態で、特定の発現パターンを示す遺伝子群を抽出したら、発現量という情報を回復してあげて、その中でも発現量が多い遺伝子と少ない遺伝子を分けることができます。数値計算だけで得られるP値より、発現がなかったところから出てきたという情報のほうが、生物学的には有意義なことが多いでしょう。
なぜ、比ではなく、対数比にするのでしょうか?
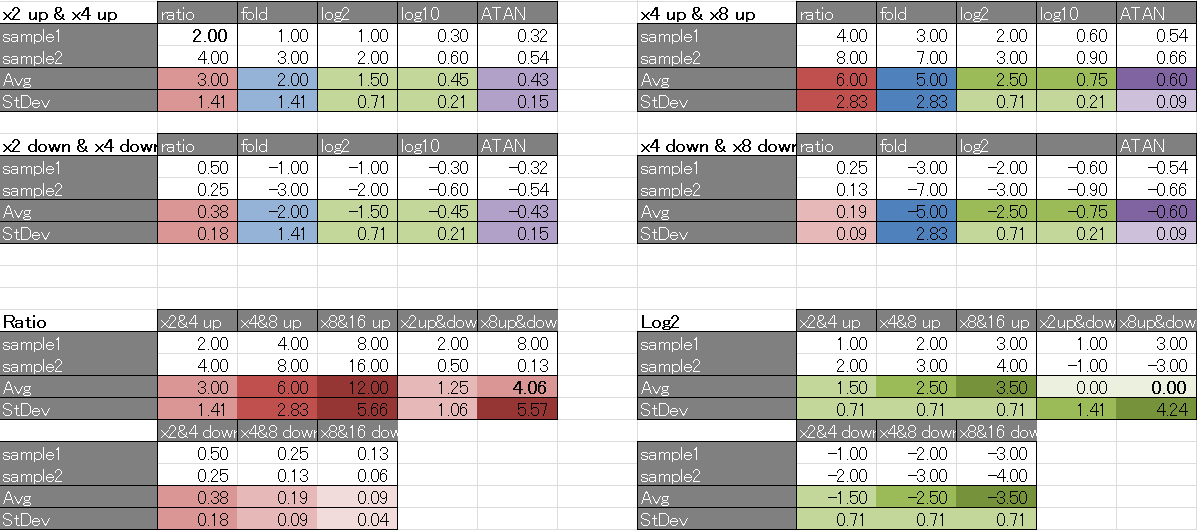
比(赤)は、増えたほうと減ったほうが対等に扱われません。増えるということは、比の値は100でも1000でも大きく変わりますが、減るということは、どれだけ減っても距離が1より大きくなることはありません。たとえば、「8倍増加(8)と8倍減少(0.125)の平均値は、4.06倍増加です」と聞いたら何かおかしいとおもいませんか?
増加と減少を対等に扱うにはいくつかの方法があります(青、緑、紫)。これらは、どれも増加と減少を対等に扱っています。ですから、どれでも8倍増加と8倍減少の平均値は「変化なし」となります。これは、わたしたちの常識と一致します。これらの違いは、増加分と分散に現れます。
対数は、この中でもよく特性が知られていて、よく使われている手法です。対数の底は2でも10でもeでも、クラスタリングや検定の結果は同じになるので、なんでもかまわないのですが、よく使われる2がわかりやすいです。
なぜ、シグナルをノーマライズするのか?
ここまで読んで、シグナルを対数比に変換する理由がわかったと思います。もう一つ前処理で大事なのが、正規化(ノーマライズ)です。
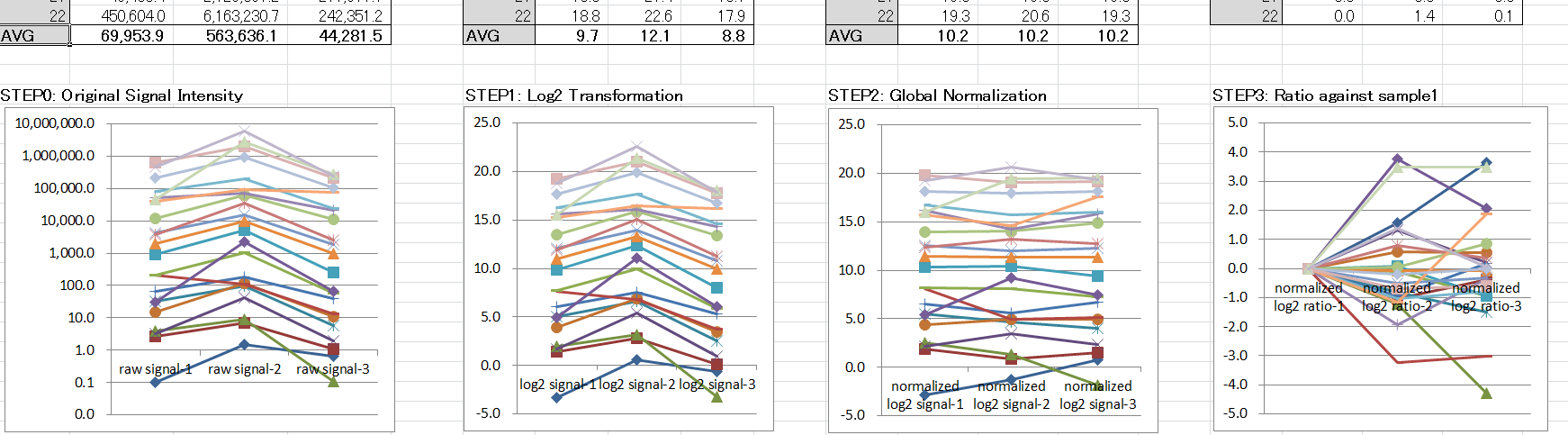
実験データを見てみると、ハイブリダイゼーションやウォッシュ、反応液の成分などの実験的な要因のわずかな差で、サンプル間にシステマティックなバイアスが生じることがあります。たとえば、そのようなバイアスが生じることを、エクセル上でシミュレーションしてみたのが、下のダウンロードリンクのものです。このエクセルシートで、3とセットすると、Sample2 では Sample1 より3倍高い値に、Sample 3では Sample 1 より3倍低い値になります。
Step1: 底が 2 の対数に変換します。この段階では、システマティックバイアスがあることがわかります。
Step2: グローバルノーマライズを適用します。サンプル間のシステマティックバイアスがキャンセルされることにより、遺伝子の本来の変動が表れます。
Step3: 比に変換します。こうすることで、どの遺伝子の発現が増加(減少)したかが、よくわかるようになります。
まとめると、前処理とは、「シグナル値」を「ノーマライズされた対数比の値」にすることです。Subio Platform 上でNormalizationをご確認ください。多くの場合、上記の3つのステップが含まれていることがわかります。
しかし、ノーマライズはいつも適用できる、あるいは適用したほうがいいとは限りません。たとえば、異なる種類の細胞を比べる時、または異なる発達ステージをくらべるとき、発現量が全体的に変わらないという前提が成立しません。このような場合は、ノーマライズを適用すべきか、すべきでないか慎重に判断しなければなりません。詳しくは、Why Subio Platform? という記事をご覧ください。
シミュレーションのエクセルシートをダウンロードする
こうしてみると、Normalizationのプロセスは単純で、エクセルでも再現できる ものであることがわかるでしょう。一度は、このシミュレーションの数式を追って、何が行われているのか具体的に理解すると、今後さまざまな応用がきくようになるでしょう。