ほとんどの方が発現差のある遺伝子群(DEGs)、つまりあるグループで高発現していて、もう片方のグループで低発現しているパターンを示す遺伝子群を探そうとします。しかし、このような遺伝子が原因遺伝子であるのは、下記のモデルのうち single factor model でしかなく、これは高度に複雑な生物現象の中では極めて稀だと思います。それ以外の下記のようなシンプルなモデルにおいて、遺伝子発現がどのようなパターンを示すかを頭に入れておくことは有用ではないかと思います。
1因子のモデル
モデル1: Single Factor Model
たとえば、原因遺伝子が一つで、その遺伝子の発現が高い(低い)ときに発症するモデルを考えてみましょう。このような発現パターンは、発現データを解析するかがた思い描く典型的なパターンですが、生物学的には極めて稀なケースだと考えられます。
健常群と病気群は、散布図上で一次元的に分離されます。
2因子のモデル
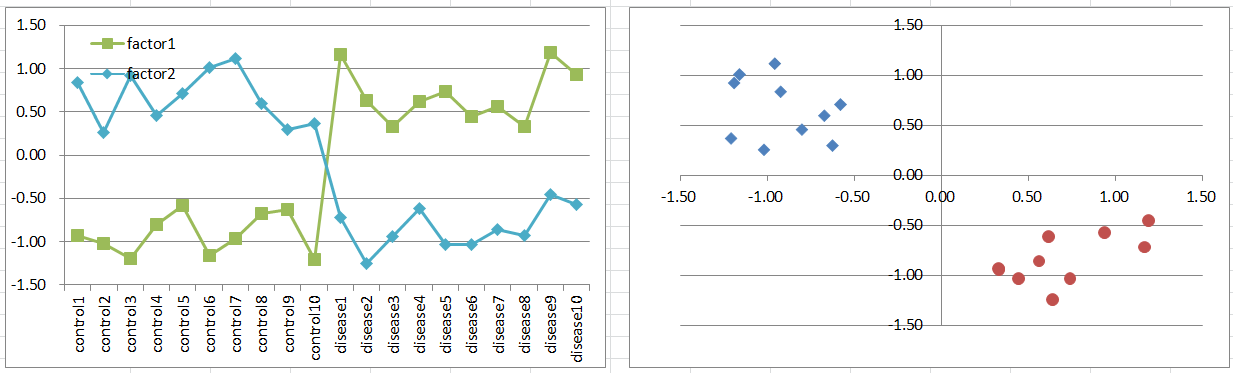
モデル2: Two Factors Model
二つ原因遺伝子があって、どちらか一方でも高発現していたら発症するモデルを考えてみましょう。これらの遺伝子群はホモログかもしれませんし、同一パスウェイへの異なるインプットかもしれません。もしもメンデルのように数えることができるなら、健常:病気の比率は1:3となるでしょう。
病気群における原因遺伝子の発現レベルは、二山型になるので、T検定が想定するような正規分布を期待できません。それでもT検定によるP値が小さいものを集めると、このような遺伝子が含まれる可能性が高いというのがおもしろいところで、原理的に不適当でも、実践的にT検定が有用である理由です。ただしこのとき、P値自体にそれほど意味があるわけではありません。
二つの原因遺伝子を軸に取った散布図上で、健常群は一つの象限にあって、残りの3つの象限に病気群が散らばります。
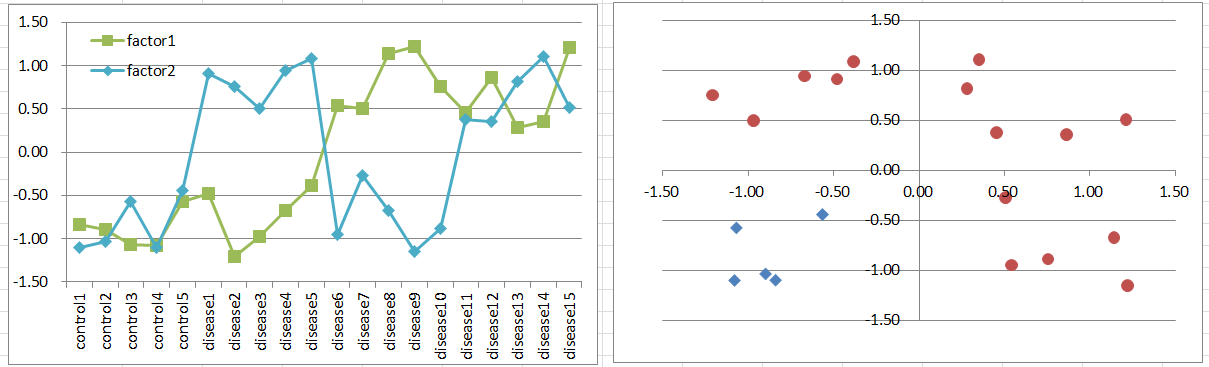
モデル3: Co-factors Model
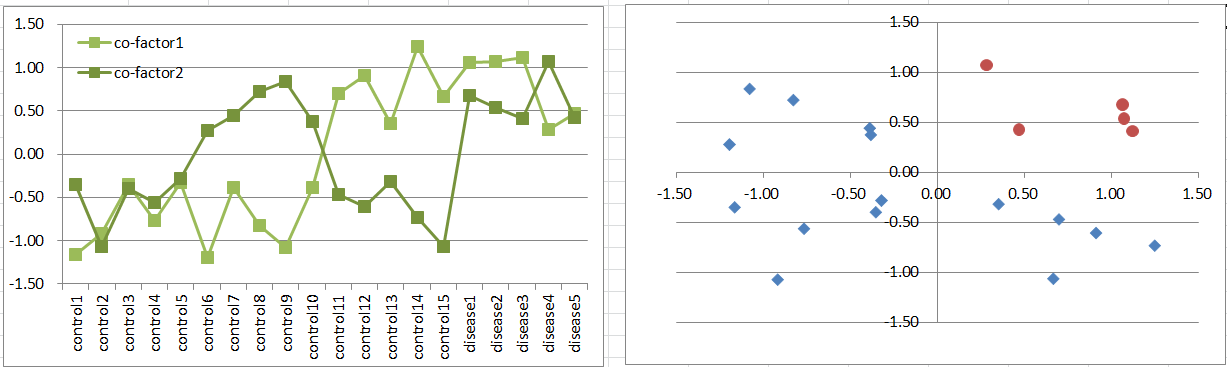
二つの原因遺伝子があるけども、その二つが同時に高発現を示すときに発症するモデルを考えてみましょう。これも生物学的にはシンプルであり、現実的にありそうなモデルです。
これもモデル2に似ているのですが、健常:病気の比率が3:1となり、モデル2とは逆転します。ここでもT検定の前提は崩れているのですが、それでもなお有用であるという点で同じです。
二つの原因遺伝子を軸に取った散布図上で、健常群は3つの象限に散らばって、残りの1つの象限に病気群が集まります。
モデル4: Factor + Inhibitor Model
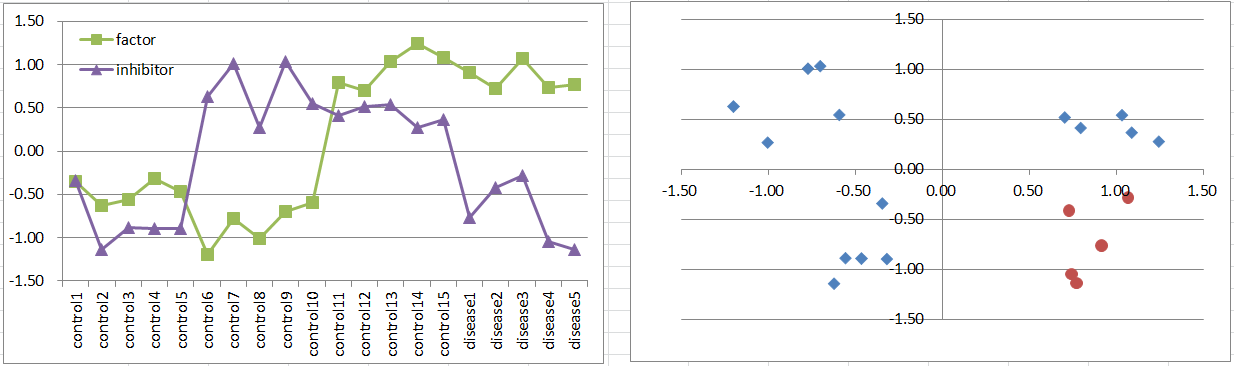
原因遺伝子が一つあって、これが高発現することで発症するのですが、抑制遺伝子が同時に高発現しているときには発症しないモデルです。これも生物学的にはシンプルかつ現実的なモデルです。
これはモデル3と、発現パターンも散布図における分布もよく似ています。
Model5: 2 Balancers Model
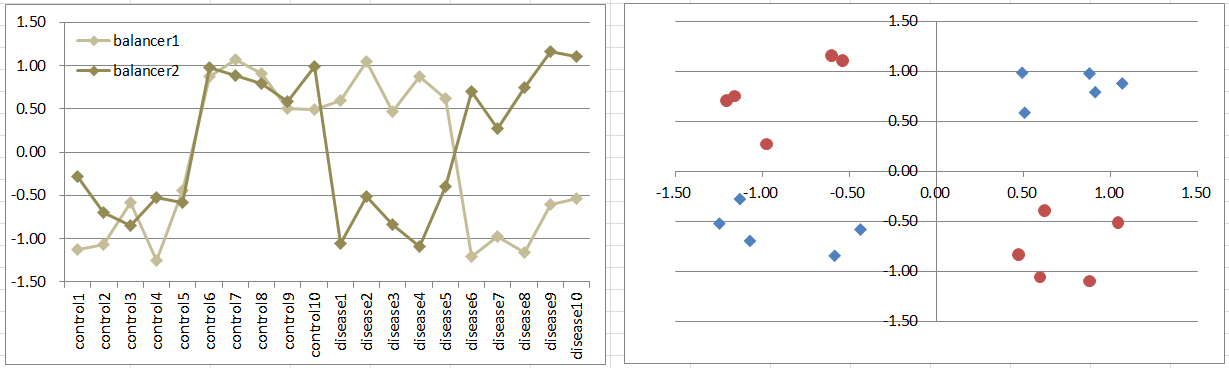
二つの原因遺伝子の発現量のバランスが崩れたときに発症するモデルを考えてみましょう。たとえば、あるパスウェイに拮抗する2つの入力シグナルがあって、これらのバランスが取れているときはその量に関係なく正常に働いているのが、どちらかの遺伝子が過剰になったときに異常な状態に陥るというものです。ダイナミクスのシンプルなモデルといえるでしょう。
健常群と正常群は、2つの遺伝子を軸に取った散布図において、互いに交差する象限に分布します。このような遺伝子は、DEGに似たパターンではないので、T検定によって抽出することはできないと考えられます。しかし、それでもシミュレーションしてみると、かなりの確率でP値が0.05を下回ります。実際にエクセル上でシミュレーションをしてみたい方は、こちらから Download してみてください。
モデル2~5は、原因は異なるのに結果として似た特徴があることに注意してください。この次は、因子の数を3つに拡大したモデルです。
3因子のモデル
モデル6: 2 Factors + Inhibitor Model
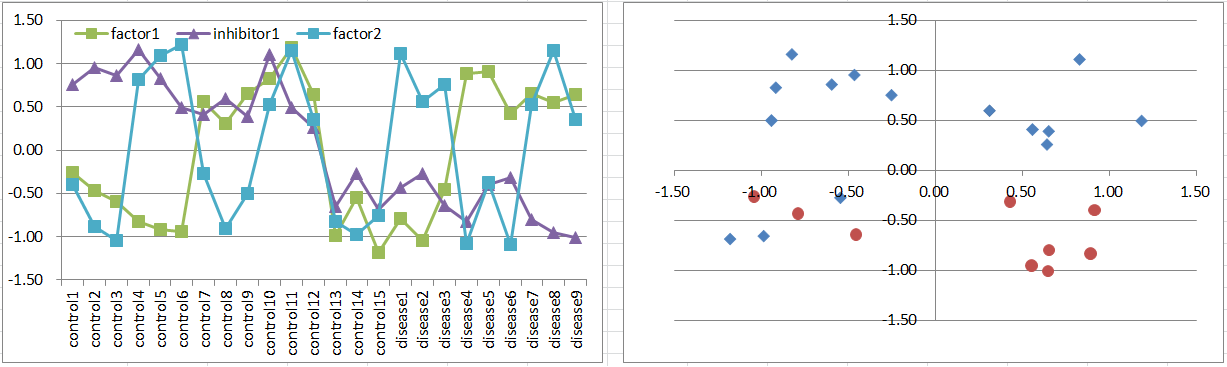
2つの原因遺伝子のうち、いずれかが高発現することにより発症するのですが、抑制遺伝子が同時に高発現しているときは発症が抑制されるもでるです。
因子が一つ加わっただけですが、パターンはぐっと複雑になります。統計検定によって抑制遺伝子は抽出することができそうですが、原因遺伝子のほうがかなり難しくなりそうです。
3次元の空間の中で、病気群は3/8の象限に分布します。
モデル7: Factor + Co-inhibitors Model
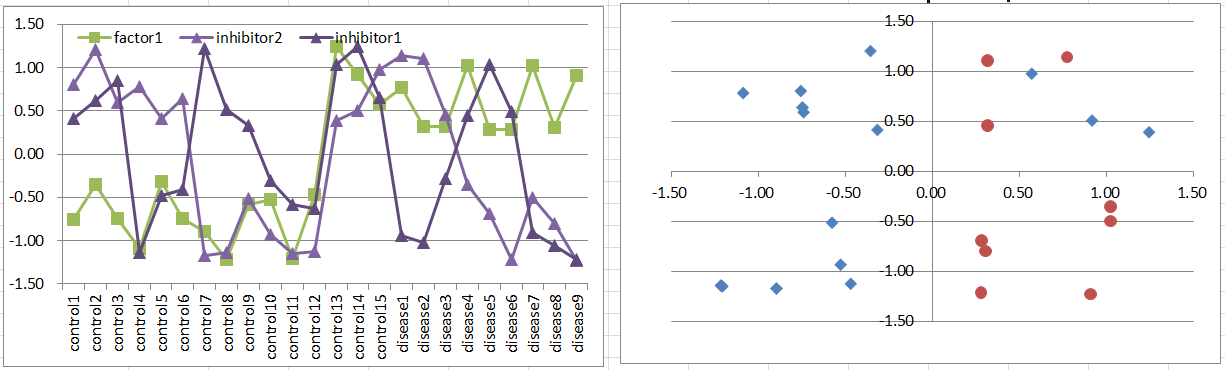
逆に、原因遺伝子が一つだけ存在し、抑制遺伝子は同時に二つ高発現していないと病気を抑制できない場合を考えてみましょう。
このモデルのパターンは、モデル6によく似ています。
モデル8: Co-factors + Inhibitor Model
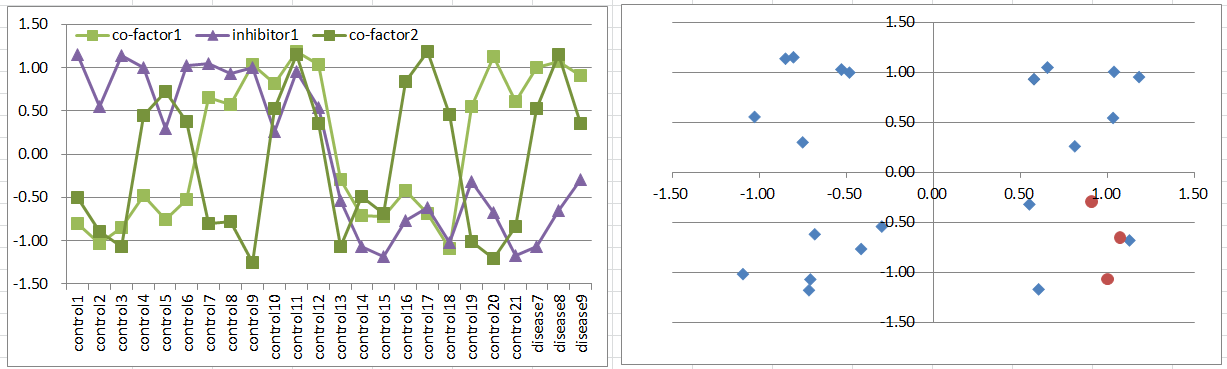
二つの原因遺伝子の両方が高発現しているときに発症しますが、たった一つの抑制遺伝子によって発症を抑制できるモデルを考えてみましょう。
この場合、病気はモデル6やモデル7よりも稀で、3次元空間で1/8象限にしか存在しません。
Model9: Factor + 2 Inhibitor Model
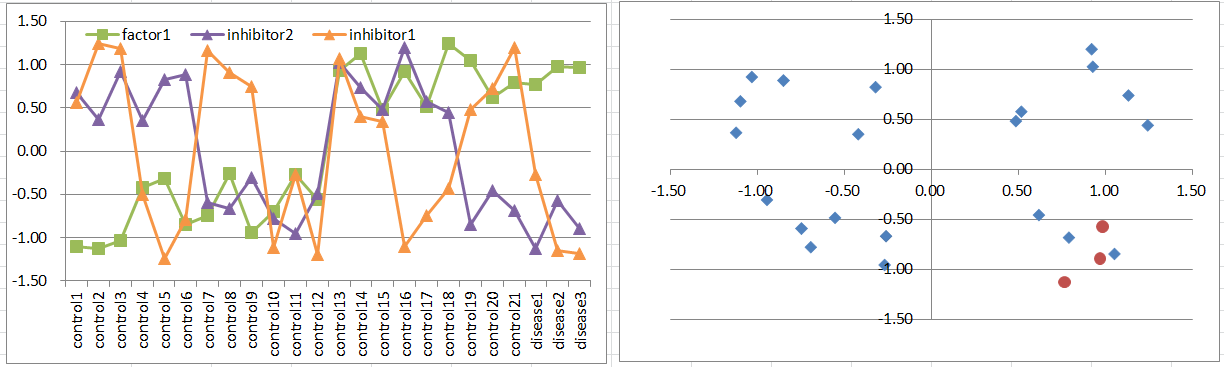
原因遺伝子が一つで、これが高発現するときに発症しますが、抑制遺伝子は二つあり、これは両方高発現していないと発症を抑制できないモデルを考えてみましょう。
これはモデル8とよく似たパターンとなります。
Model10: 3 Balancers Model
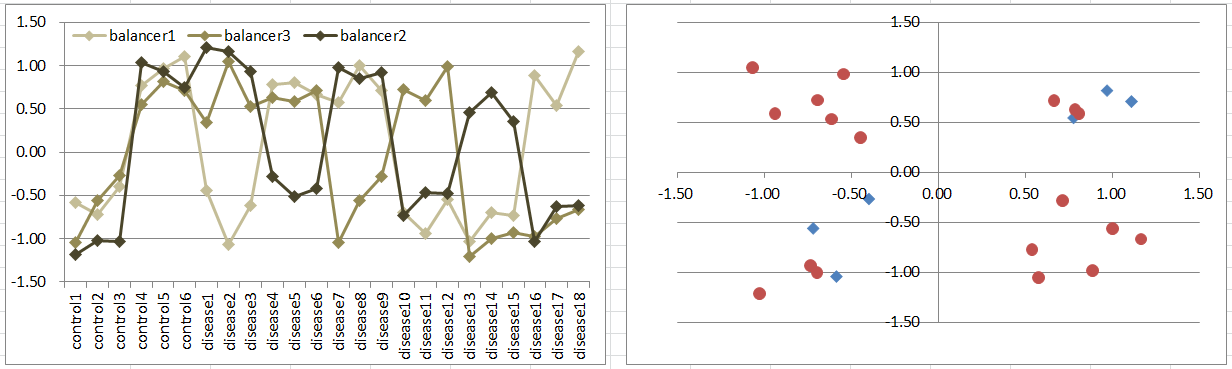
最後に3つのバランサー遺伝子のモデルを考えてみます。これらの遺伝子の発現量が拮抗しているときは正常なのですが、バランスが崩れると発症するというモデルです。
これはモデル5に似ていますが、3次元空間の対角線の象限に健常群が並んで存在します。
Download the Excel file of this simulation from DropBox.
Conclusion
原因は違えど、パターンはモデル3と4、6と7、8と9、5と10でそれぞれ似ていることがわかります。つまり、表面に現れる発現パターンは、背後にある発病メカニズムの数よりも少ない可能性があります。がんのような複雑な病気のデータを扱っているときに、DEGsが見つからないと嘆く必要はないのです。このようないくつかのパターンを頭に入れながらデータを見て、見えているパターンから背後にありそうなメカニズムを考えるということが必要ではないかと思います。加えて、現実ではモデルのように、健常と病気のサンプルはバランスよく集まらず、病気サンプルに偏りがちです。さまざまな条件を考えながら、データからメカニズムを推量しなければいけないのです。コンピューターに推量をやらせても、可能性のある擬陽性のモデルはごまんと出てきて、どれが当たりなのかを判定することはできません。データ化できない事情にも精通している生物学者にしかできないと思うのです。
DEGs を探すという行為は、下流の遺伝子群の抽出には有効ですが、原因遺伝子を探すという意味ではきわめて稀なケースだと思われます。現実とくらべるととても単純化された上記のようなモデルでも、原因遺伝子が DEGs のようなパターンを示すことはほとんどありません。それでもシンプルなT検定はツールとして有用です。それは、緩めのP値がゆるめ(0.05 ~ 0.1)のところに原因遺伝子の候補が集まることが予想されるからです。P値が低い(きわめて有意)ところに集まるわけではないというところがポイントです。