はじめに
Subio Platform を使えば、TCGAのRNA-SeqとDNAメチル化アレイのデータを簡単にインポートできます。(インポートの操作は、"TCGAのRNA-Seqデータをインポートする" または "TCGAのDNAメチル化データをインポートして解析する" のチュートリアルをご覧ください。) 遺伝子発現とDNAメチル化を統合解析する実践的なケーススタディを始めましょう。
それでは、TCGA-LIHC (Liver Hepatocellular Carcinoma) を例にとって、まずはDNAメチル化データの概要を見ていきましょう。RNA-Seqで測定されている遺伝子のTSSから4kbp以内にあるメチル化サイトを抽出して調べました。
このDNAメチル化のデータはIllumina社のInfinium HumanMethylation450 BeadChipにより測定されています。このアレイの設計では、TSSの上流200bpおよび1500bp以内、そしてCpGアイランドにあるメチル化サイトを測定するためのプローブが集中的に搭載されており、ゲノム上のメチル化サイトを均等に搭載しているわけではありません。したがってTSSプロットでは、TSSの近傍には点が濃く見えますが、これは生物学的事実を反映したものではないかもしれないことにご注意ください。
TCGA-LIHC メチル化データの概要
Fig 1はbeta valueのTSS Plotです。Beta valueは、メチル化の度合いを0(メチル化されていない)から1(メチル化されている)で表します。
CpG アイランドでは、ほとんどのサイトが脱メチル化状態を維持されていますが、TSS近傍の一部のサイトでは高メチル化状態になっていることが分かります。一方、CpGアイランド以外のサイトの多くはメチル化されていますが、TSSの周辺(1~1.5kbp以内)に限ると脱メチル化しているものが多いです。
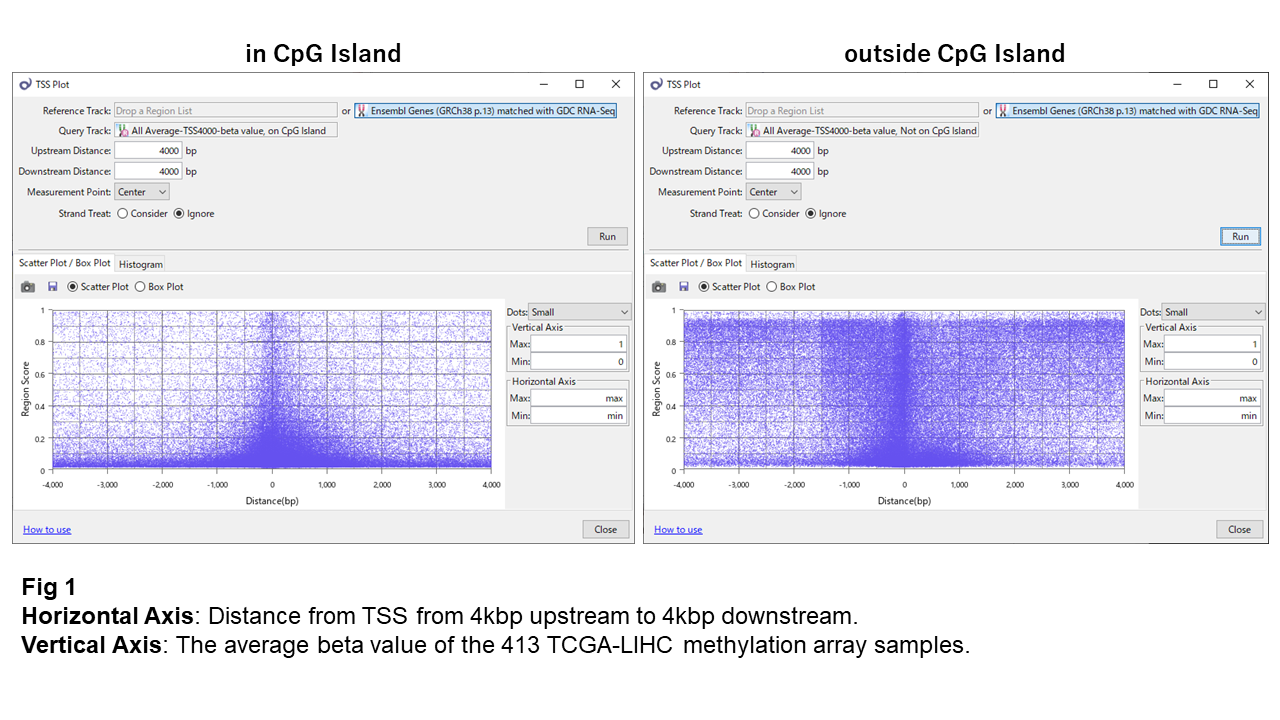
Fig 2 のTSS Plotは、Normal vs. Tumor のbeta valueの変化量を縦軸に取っています。縦軸上の正数はTumorでよりメチル化されたこと、負数はTumorで脱メチル化されたことを表します。
CpGアイランドでは、メチル化状態が変化しないよう厳しく制御されているようです。しかし、TSSの近傍では一部のサイトでbeta valueが変動しています。そのうち大部分はメチル化が増加する方向に変化しています。一方、CpGアイランド以外のサイトでも多くはメチル化状態が変化しないよう保存されていますが、CpGアイランドに比べると制御が緩いかもしれません。そして、CpGアイランドでは、多くが脱メチル化の方向に変化しています。
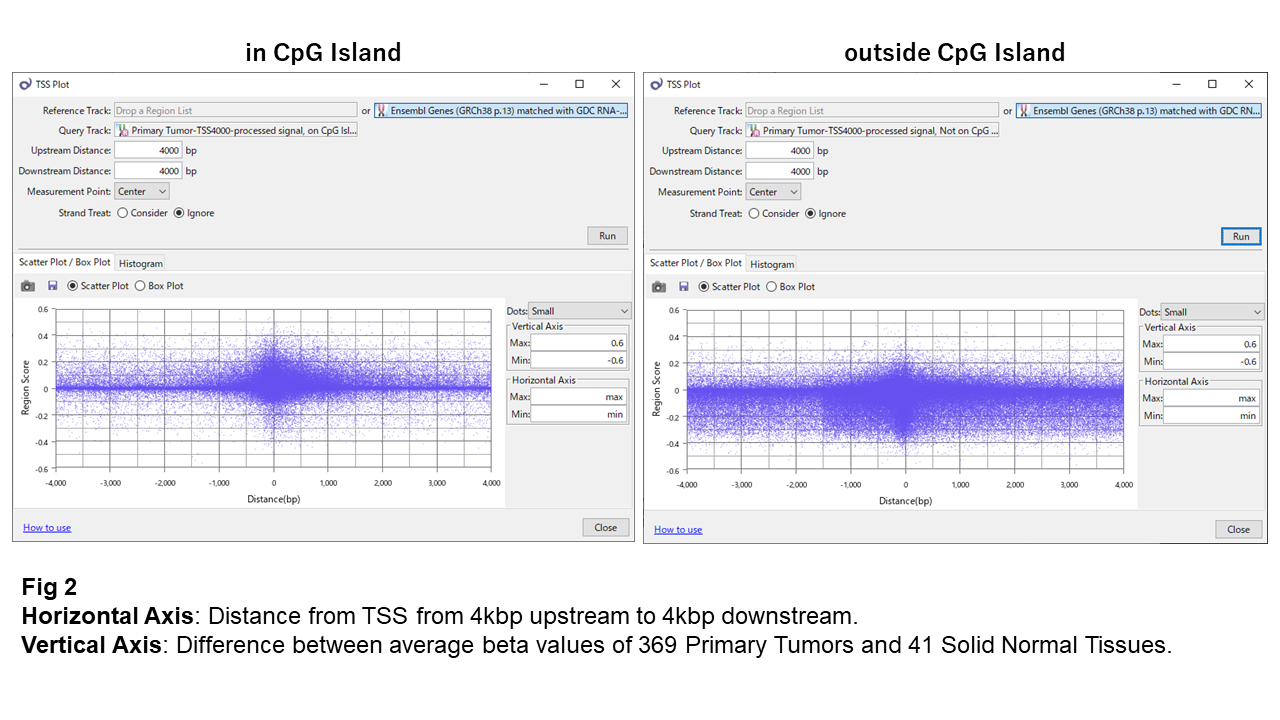
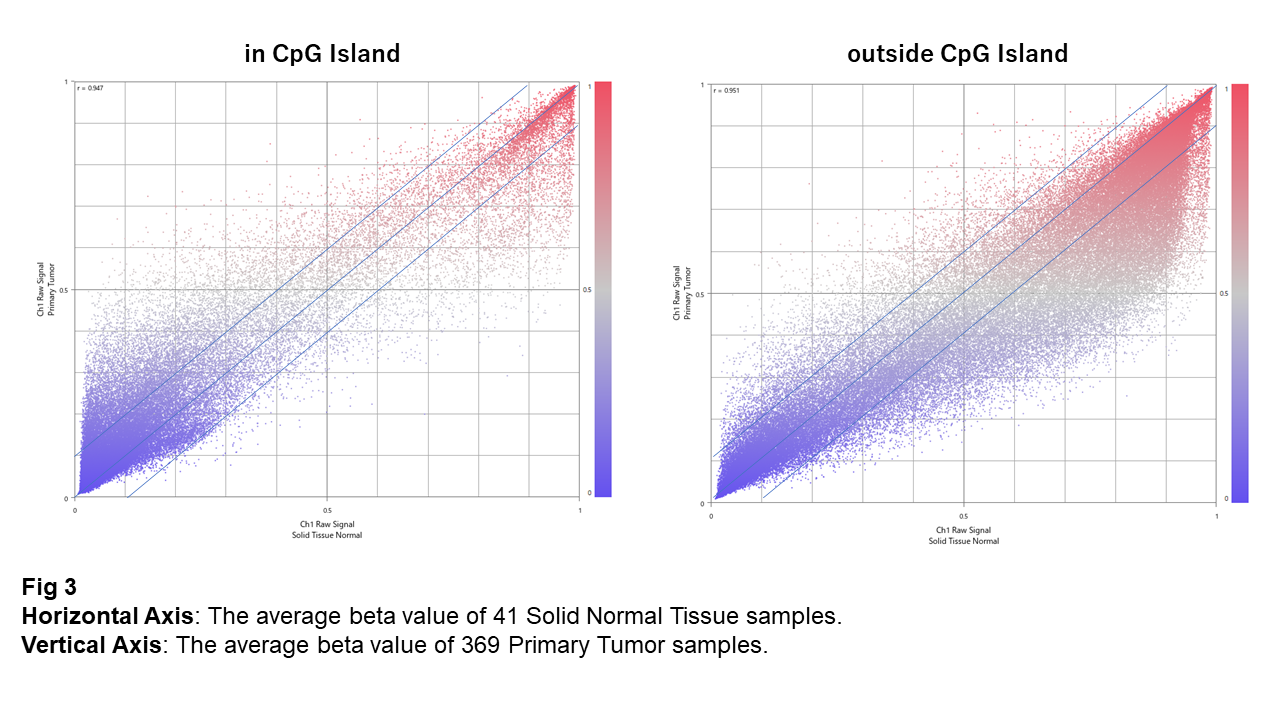
Fig 3はNormal vs. Tumorのbeta valueの平均値を比較した散布図です。CpGアイランドでは、脱メチル化状態から一部のサイトでメチル化が亢進していることが分かります。一方、CpGアイランド以外のサイトでは、メチル化の高いサイトのうち一部がTumorで脱メチル化の方向に変化していることが分かります。
メチル化と発現パターンの相関
ここからが本題です。上記のような特徴を持つメチル化状態の変化が、どのくらい近傍遺伝子の発現量に影響を与えているのでしょうか。
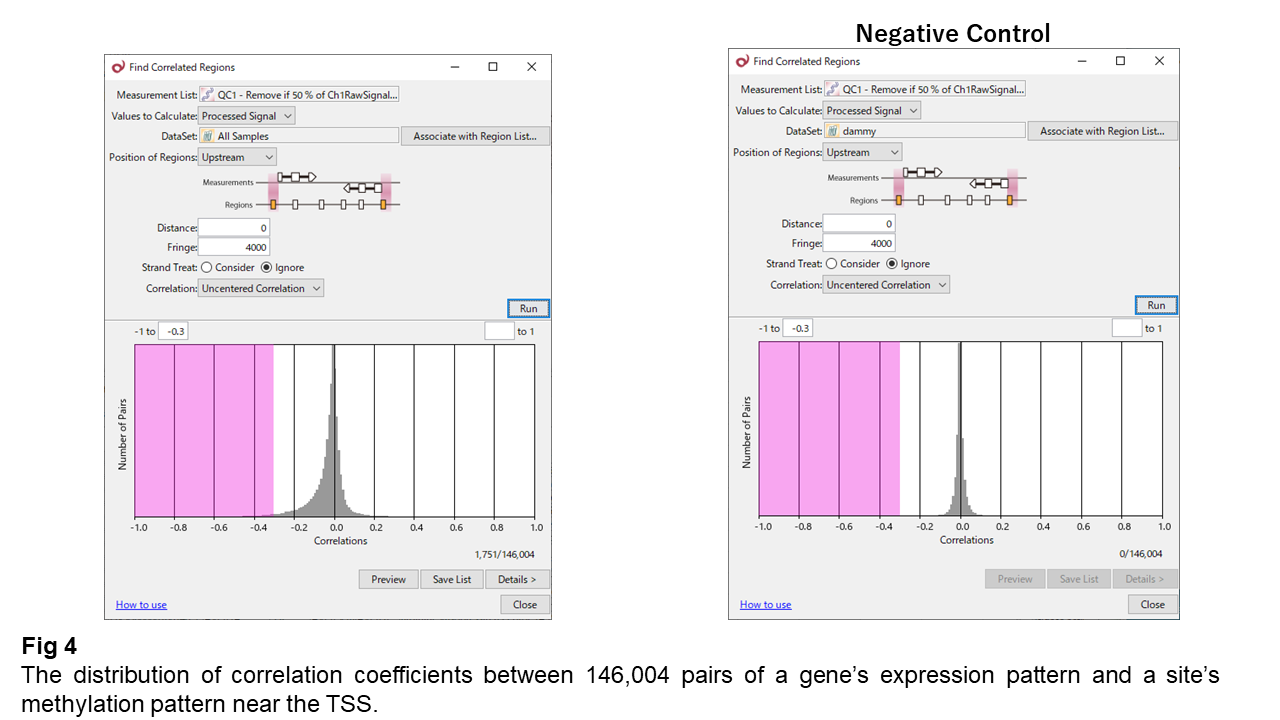
Fig 4は、メチル化の変動パターンと、近傍遺伝子の発現変動パターンの相関係数の分布を表しています。ほとんどの組み合わせで相関係数は0付近に収束しており、二つの変動パターンはほとんど無相関ということが分かります。しかし、分布形は左に傾いており、一部の組み合わせでは弱い逆相関を示すようです。
RNA-Seqとメチル化アレイのサンプルの順序を適当に入れ替えて計算すると逆相関への偏りが消失する(Negative Control)ので、この逆相関は、たとえ弱くとも有意と考えられます。ここでは、逆相関を示す組み合わせとして、相関係数が-0.3以下のものを抽出しました。
縦軸に相関係数をとったTSSプロット(Fig 5)を見ると、逆相関を示すサイトはTSSのごく近く(300bp以内)に集中していて、TSSから離れるとほとんどなくなります。逆相関を示すサイトの数は、CpGアイランドに1,007個、CpGアイランド外に715個ありました。数の上ではCpGアイランドのほうが多いですが、もともとプローブ数に偏りがあるため、-0.3を下回る割合を比べると1.4%と1.3%となり、ほとんど差がありませんでした。
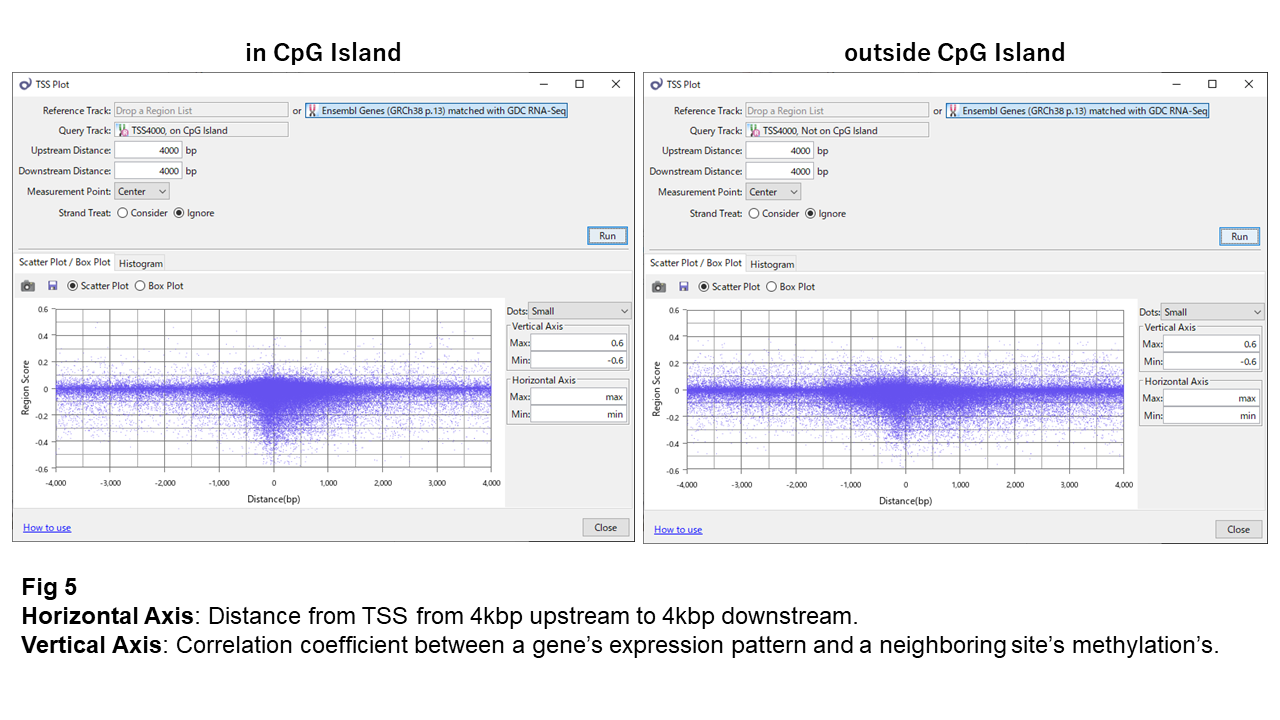
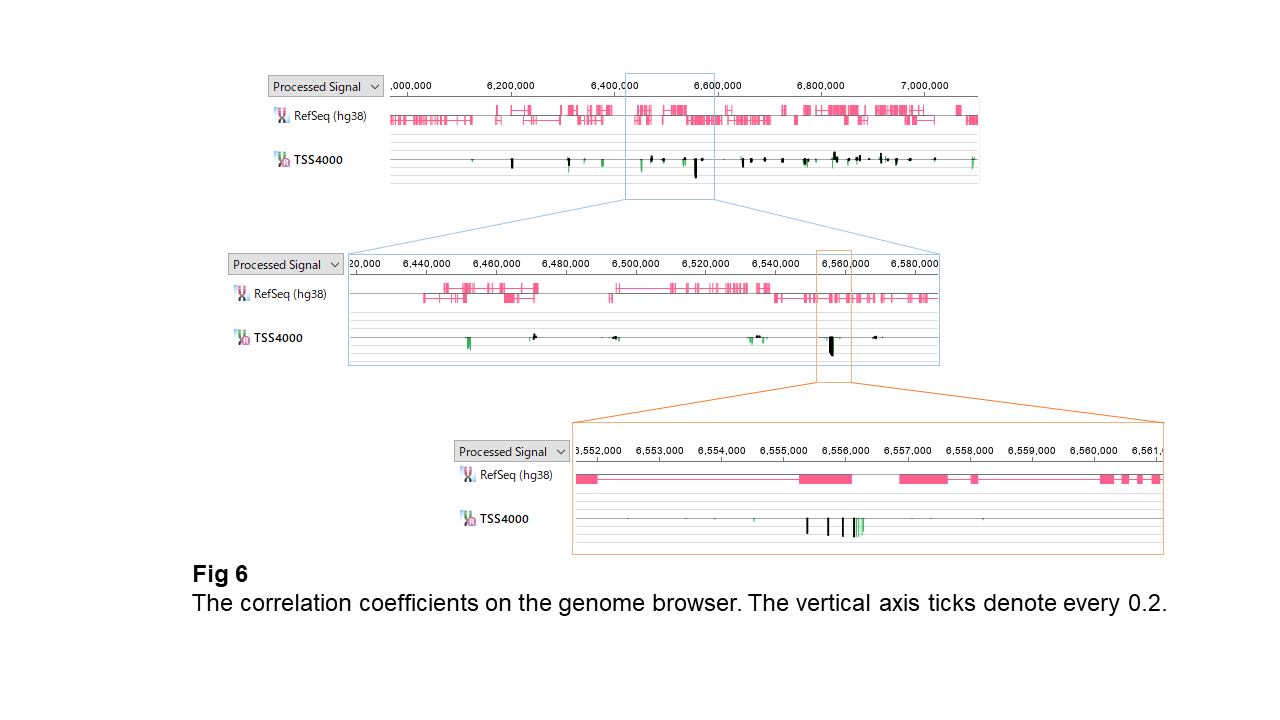
Fig 6はゲノムブラウザー上に、遺伝子とそのTSS周辺4kbpにあるメチル化サイト、そして各メチル化サイトの位置には、メチル化パターンと近傍遺伝子の発現パターンの相関係数を棒グラフで表示したものです。黒い棒グラフはCpGアイランド、緑のはCpGアイランド外のサイトです。ほとんどの遺伝子のTSS周辺では相関係数が0に近い値ですが、ときどき相関係数が-0.2の目盛線を下回ることがあります。そして、それはCpGアイランドの時も、CpGアイランドではないときもあります。隣接するメチル化サイトの相関係数がだいたい同じなのも注目に値します。
CpGアイランドのメチル化状態と、近傍遺伝子の発現量の関係
ここまで個別のメチル化サイトについて見てきましたが、ここからはCpGアイランド単位で見ていきましょう。そのために、まずはCpGアイランドごとにベータ値の平均値を求め、TSSから500bp以内にCpGアイランドを持つTCGA-LIHC RNA-Seqで発現量を測定されたタンパク質コーディング遺伝子と組み合わせたところ、11,035対のペアができました。
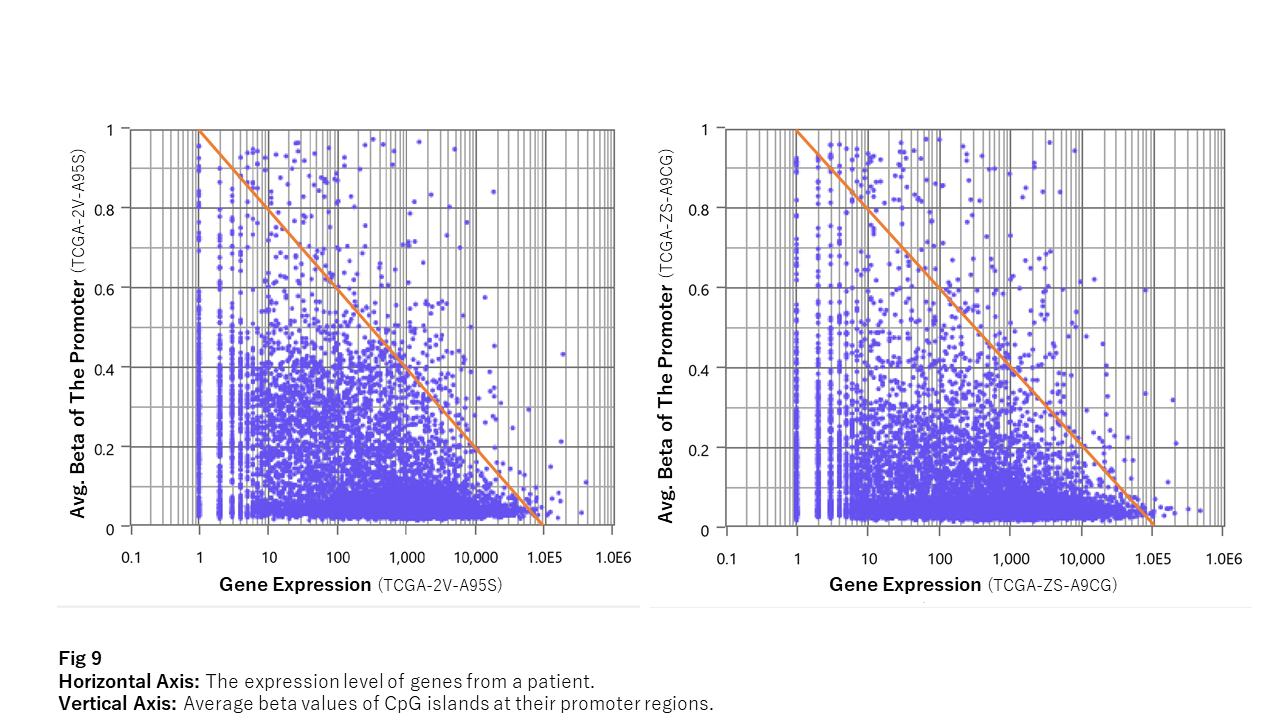
Fig 9 は、CpGアイランドの平均ベータ値と近傍遺伝子の発現量の関係を散布図で示したものです。ここでは、例としてTCGA-2V-A95SとTCGA-ZS-A9CGの2サンプルについてだけ図にしています。点の集りが三角形を形成していることから、プロモーター領域のメチル化レベルにより近傍遺伝子の発現量が抑制されていることが示唆されます。
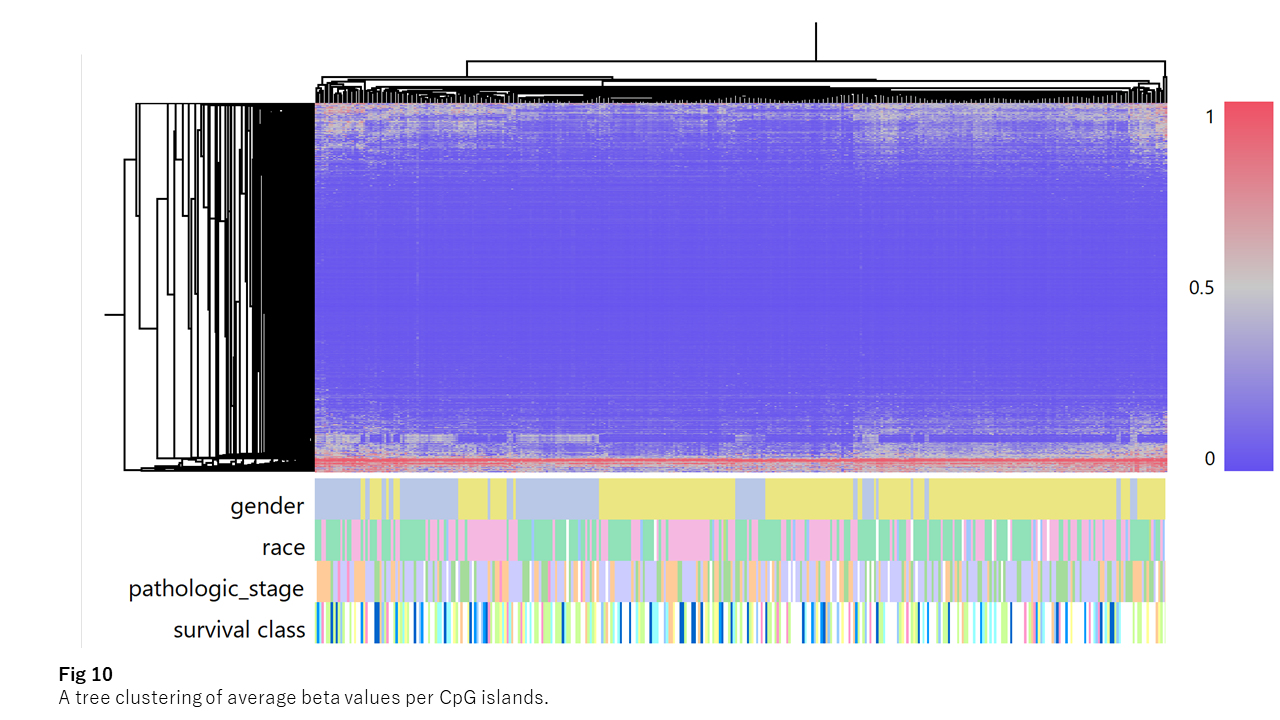
これらCpGアイランドの平均ベータ値を、全患者について俯瞰したのがFig 10です。大部分の遺伝子のCpGアイランドは、すべての患者さんで脱メチル化状態に保たれていることが分かります。ごく一部ですが、CpGアイランドがすべての患者さんで高いメチル化レベルに保たれている遺伝子もあります。これらを除く約4000個のCpGアイランドでは、平均ベータ値が患者間で変動しており、近傍遺伝子の発現量に影響を与えている可能性があると考えられます。(Fig 10)
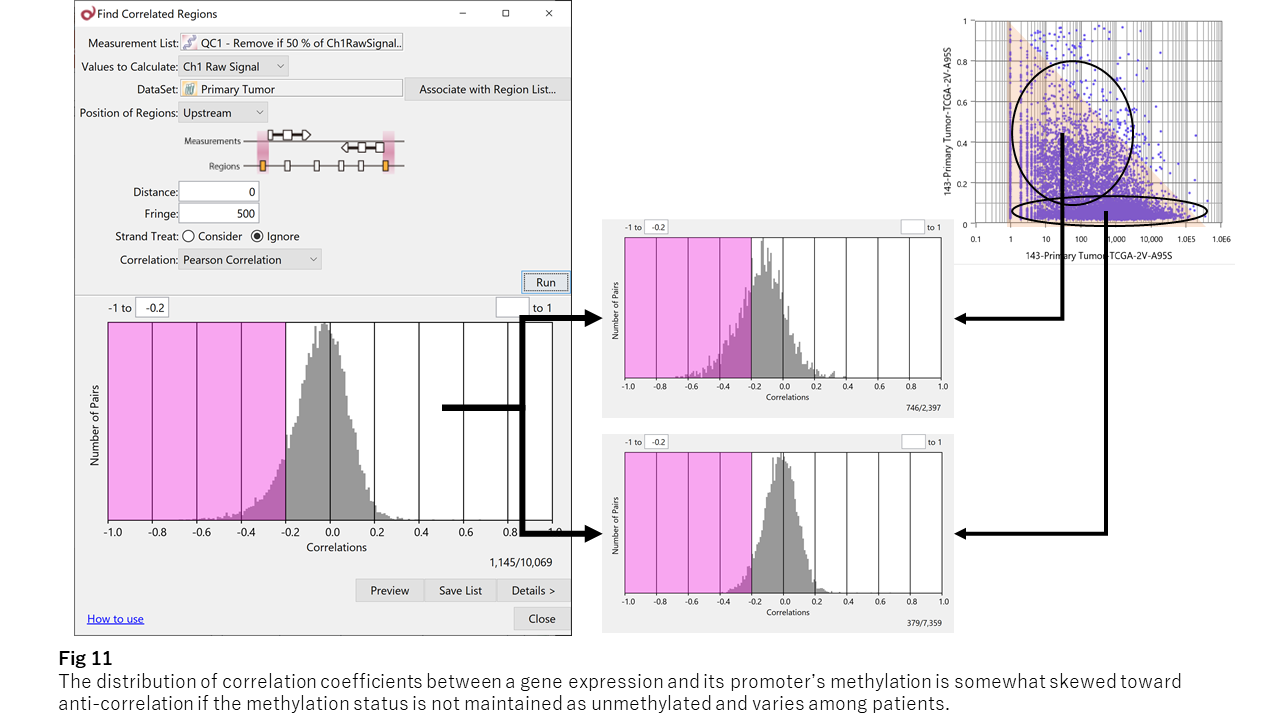
Fig 11 は、CpGアイランドの平均ベータ値の変動パターンと近傍遺伝子の発現パターンの相関係数の分布を示したものです。ピークは無相関を示す0付近にありますが、全体としては逆相関の方向に偏りが見られます。プロモーターのメチル化状態が近傍遺伝子の発現制御に関わっていることを示唆していると考えられます。
Fig 10では、大部分の遺伝子でプロモータが脱メチル化状態に保たれていることが示されました。そして、予想通りこれらのプロモーターと近傍遺伝子の発現量の間に関係は見られませんでした。一方、メチル化状態が変動するプロモーターでは、近傍遺伝子の発現パターンと弱い逆相関を持つことが示されました。
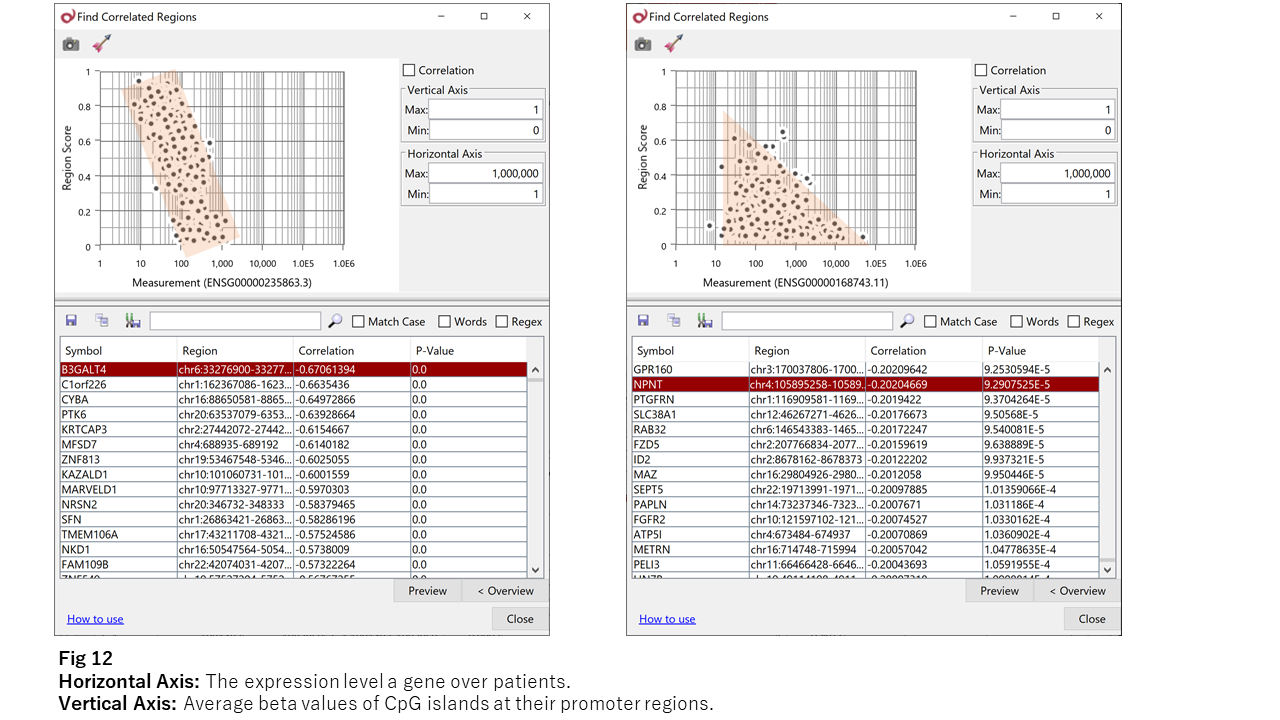
Fig 11で示された通り、たとえばB3GALT4のような強い逆相関を示す遺伝子(Fig 12の左図)はほとんどありません。それよりも、たとえばNPNTのように点の集まりが三角形になる遺伝子が多く見られます。(Fig 12 の右図)
つまり、プロモーターのメチル化度の上昇が近傍遺伝子の発現を抑制するのに対し、メチル化度の減退かならずしも発現量を増加させないと考えられます。これが、弱い逆相関(相関係数が -0.2 から -0.4)が主に観測される理由ではないでしょうか。
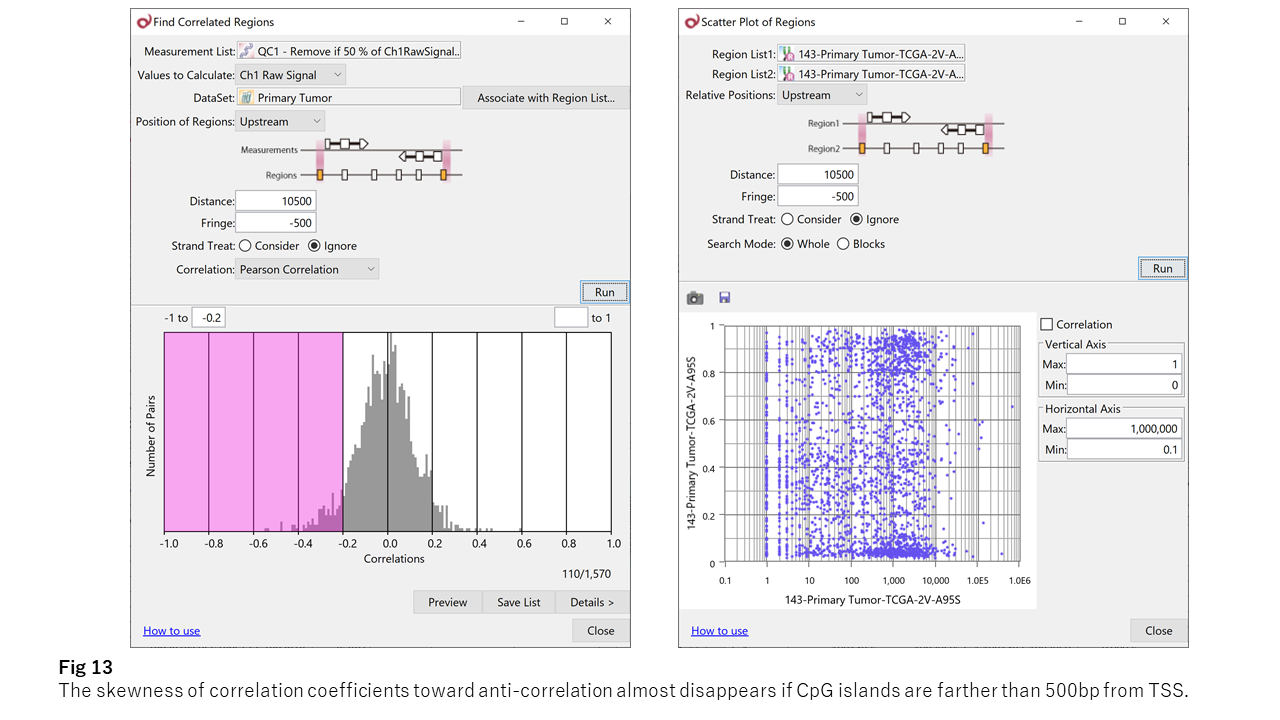
ところで、上記の逆相関は、TSSから500bp以内のプロモーター領域に特異的な現象です。Fig 13で示す通り、TSSから500bp以上離れたCpGアイランドのメチル化パターンと発現パターンの間には、逆相関の関係は見られなくなります。