GSE164898 は、10X Genomics のプロトコルで測定されたデータです。組織から数千の細胞に分けて、細胞ごとの発現量を出してくれるそうですが、1細胞あたりのリード数は数万程度しかありません。 RNA-Seqのダイナミックレンジはリード数に依存します。リード数が数万しかなければ、非常に発現が高い数十個の遺伝子の発現量しか捉えられないだろうと予想されます。それでは、実際にデータを見てみましょう。
Subio Platformを使ってデータをきちんと視覚化して見れば明らかなのですが、まず、細胞ごとのダイナミックレンジが大きく異なっています。下図の左はダイナミックレンジが最も狭いサンプル群から2つ、右はダイナミックレンジがもっとも広いサンプル群から2つを選んで、散布図を描いたものです。このようなダイナミックレンジの違いが発現プロファイルの違いの主な要因となっているのであって、論文で主張しているような生物学的な違いを反映しているのではありません。
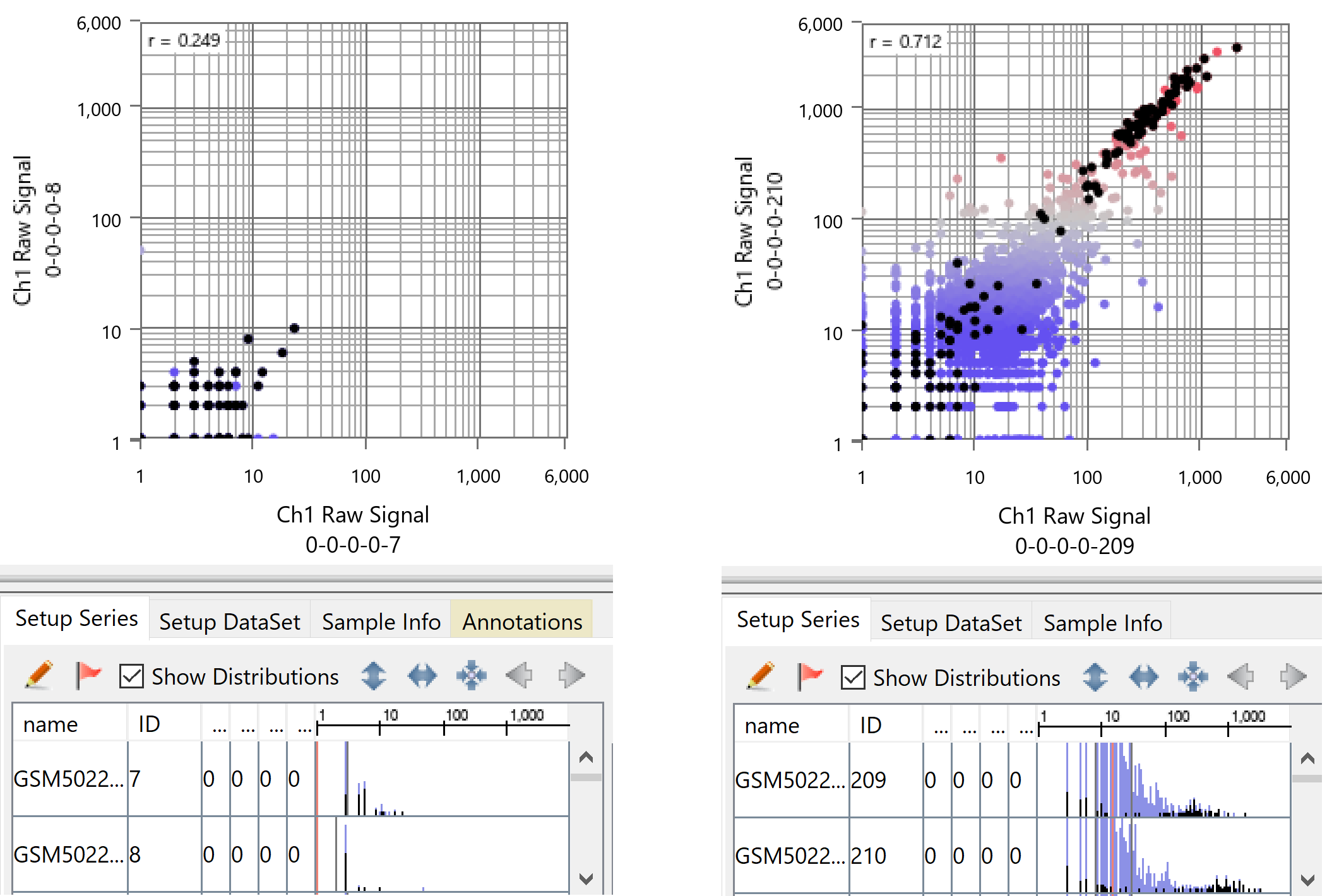
また、散布図からわかるように、countの値が10から40のあたりにシグナル領域とノイズ領域の境があります。左のサンプルでは測定値はすべてノイズ領域にあり、右のサンプルでもシグナル領域にある遺伝子は300個程度です。ノイズ領域では、測定されたcount値が信頼できないだけでなく、検出されるかどうかも偶然です。つまり、測定されなかった遺伝子が、発現していないとは言い切れないのです。また、黒い点は、ribosomal proteinの遺伝子を表しています。 count値が1以上の遺伝子であれば、ribosomal proteinn並みに超高発現している ことが示唆されます。
Loupe Browserで作られた図を、実験生物学者は免疫染色によるFISHの画像のようなものだと誤解しがちです。しかし、これで視覚化されるのは、偶然にとらえられた超高発現遺伝子に過ぎません。これで発現していないように見える遺伝子でも、実際には高発現している可能性がまだまだ十分にあるので、高感度のFISHとはまったく別物だと理解する必要があります。
シングルセルRNA-Seqはまだ技術的に限界があり、通常のRNA-Seq並みに汎用性があるとは言い難いものです。このようにSubioでは、採用しようとしているプロトコルが研究目的に資するものかどうか、具体的に公共データベースを使ってアセスメントしたうえで採用するかどうかを判断することをお勧めしています。Subioのデータ解析サービスは、アセスメント目的でもご利用いただけます。