これは Subio Platform でRNA-Seqデータの正規化・前処理を行う手順の解説です。R/Bioconductorなどを使われている方にとっても、下記のコンセプトを理解することは有益です。
正規化と前処理の適切なやり方を見つける。
Subio Platformでは、下記のプリセットのシナリオを用意しています。データに合わせて選択し、それをベースにデータの特徴に合わせて調整していくと簡単にできます。
- Expression Microarray
- RNA-Seq (Count)
- RNA-Seq (FPKM, TPM, RPKM)
- Methylation Beta Values
- Pre-normalized Log2 Data - すでに正規化・対数比化までの処理が終わっている場合に選択します。
- Nothing - なにもしない状態に戻すときに使います。
ただ、解析の経験がないと適切な処理ができているかどうか判断するのが難しいかもしれません。 オンライントレーニング では、あなたが解析したいデータでインポート・正規化・解析までひととおり学んでいただけます。
RNA-Seqのリードカウント(Count)のテーブルを見てみると、たくさんの 0 があることに気づくでしょう。0 は「マッピングされたリードが無い」ことを示していますが、即ちその遺伝子が「発現していない」とは言えないのが、RNA-Seqのデータ解析の難しいところです。
その理由は、繰返しのある実験データを見ると明らかです。例えば、繰返しのうちあるサンプルでは値があるのに、別のサンプルでは0ということがあります。これは、発現していてもシーケンサーにキャプチャーされない可能性があることを示しています。発現している遺伝子のCountが偶然 0 となるかどうかは確率的に決まり、発現が低いほど 0 となる確率が高くなることが予想されます。実際にデータを見ていると、繰り返しサンプルの一部のCountが0となる遺伝子は、ノイズ領域に集中していることが分かります。
ここで、グループAとグループBの平均値を比較して、ある遺伝子がグループBで発現量が下がって見えたとします。しかし、よく見るとグループBのサンプルにはCountが 0 のサンプルが含まれていて、測定されたCountだけ見るとグループAとあまり変わらない、ということがありえます。「Count 0」の扱い方がまずいと、偽の発現差を検出してしまうだろうということです。これを踏まえて、データ解析をどうするかを検討してみましょう。
Countデータを解析する場合
繰り返しサンプルがある場合、そのCountを散布図で見てみると、Countの高い領域では対角線上に点が収束していて、低い領域では収束していないように見えます。これがシグナル領域とノイズ領域です。シグナル領域とノイズ領域の境界は明確ではありません。また、データセットによってもまちまちですが、だいたいCountの値が100より高いところがシグナル領域、10より低いところがノイズ領域、そして10~100の間にシグナルとノイズの境界が見られることが多いです。上記のとおり、Countsが偶然0になっている遺伝子が多いのは、ノイズ領域です。(黒点)
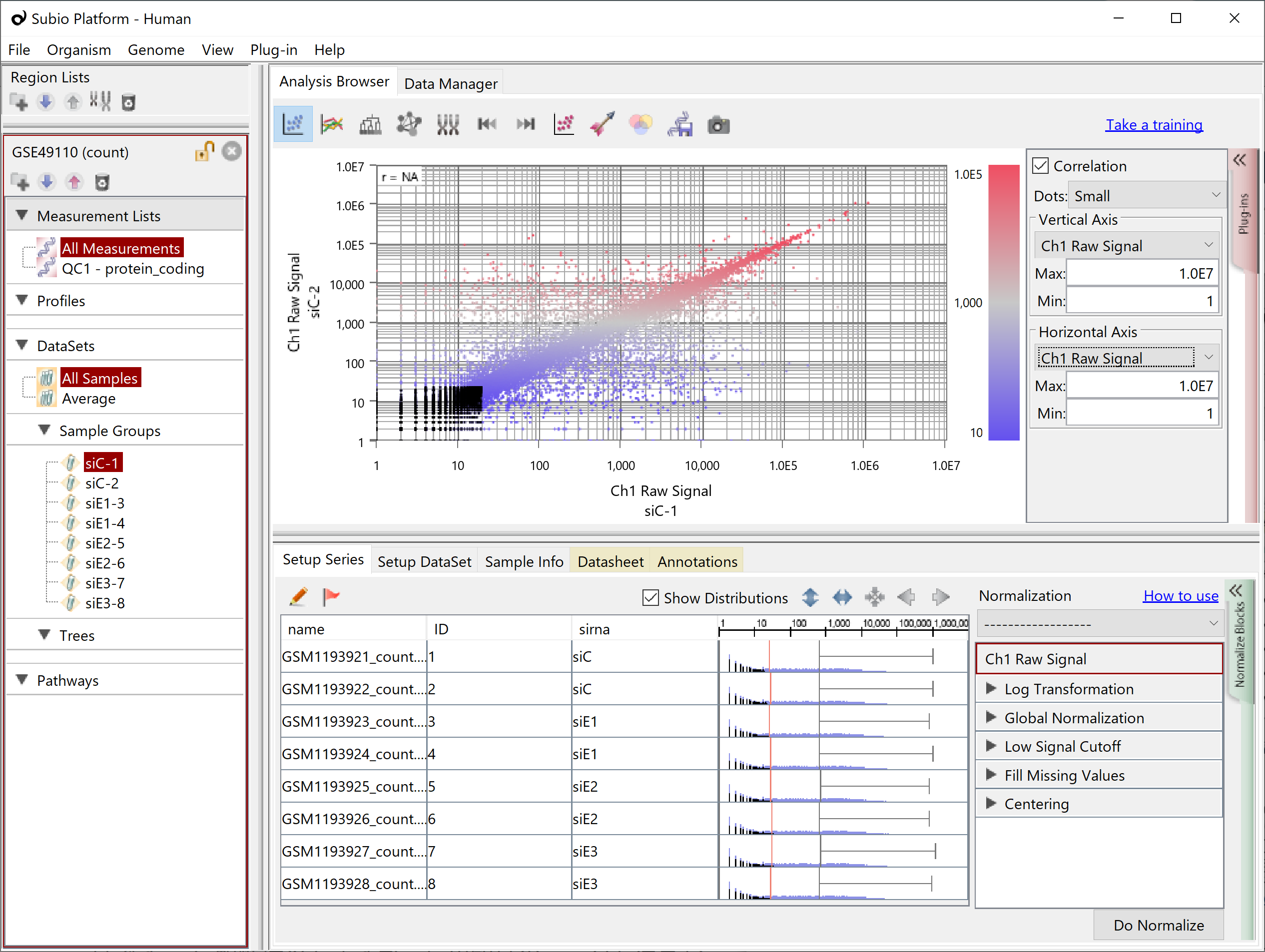
そこで、Subio Platformでは、RNA-Seq (Count) というプリセットのノーマライズシナリオを用意して、Countの値に対して次のような前処理・正規化を行います。
- Log Transformation: base 2
- Global Normalization: at 90th percentile
- Low Signal Cutoff: Replace <30 to 30
- Fill Missing Values: 4
- Centering
ステップ1で、Count 0は欠損値に置き換えられます。
ステップ2は、Global Normalizationを適用しています。これは、総リード数を100万で割って均す正規化手法と、目的も効果も同じとみなせます。しかし実際には、100万で割っても正規化が中途半端なことがありますので、たとえそのような正規化が適用されているデータに対しても、Global Normalizationを重ねて適用するほうが安全でしょう。デフォルトで90となっているpercentileの値は、ダイナミックレンジが広いほど低く、狭いほど高く調整してください。
ステップ3は、ノイズ領域の測定値が偽の発現差を検出するのを防ぐために、Countの下限値をシグナル領域の下限に合わせる措置です。
ステップ4は、Count 0だったところが欠損値になっているので、それを何かの値に置き換えます。ステップ3と同じ値か、それよりも少し低い値を設定するといいでしょう。ただし、指数で指定することに注意してください。4は、2の4乗=16の意味です。ステップ3で設定したデフォルトの下限値の約半分に相当します。
ステップ5は、全サンプルの平均値に対する比にするものです。このステップは、コントロールがある場合は、Ratio to Control Samplesブロックに置き換えることができます。
以上の計算をエクセルで再現したファイルを用意しましたので、詳しくお知りになりたい方はダウンロードしてみてください。
TPM・FPKM・RPKMデータを解析する場合
TPM・FPKM・RPKMデータを解析するのは、Countの場合よりも厄介です。なぜなら、ノイズ領域がシグナル領域混じりあって広がっている(黒点)ので、0を何らかの値に置換すると、冒頭の偽の発現差を検出する問題が顕在化してしまうからです。
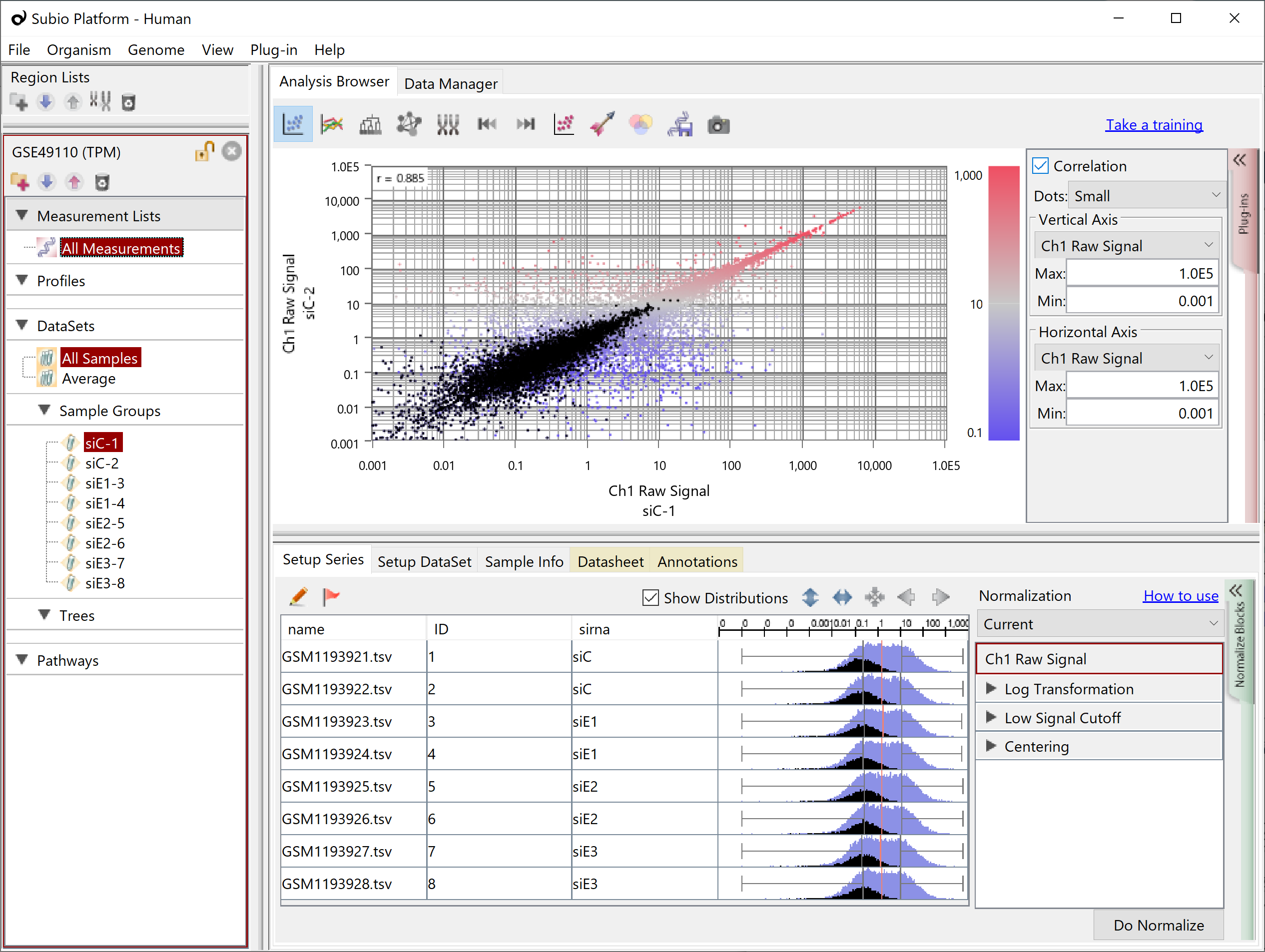
それでは、Subio Platformのプリセット・ノーマライズ・シナリオ RNA-Seq (TPM, FPKM, RPKM) を見てみましょう。
- Log Transformation: base 2
- Global Normalization: at 90th percentile
- Low Signal Cutoff: Replace <0.01 to 0.01
- Centering
ステップ2まではRNA-Seq (Count) のシナリオと同じですので、そちらの説明を参照してください。
ステップ3は、下限値を設定するものですが、Countの場合と異なり、ノイズ領域の影響をキャンセルすることはできません。下限値を設定する意味は最低限でしかありません。
Countの場合にはここでFill Missing Valuesブロックを適用していましたが、TPM・FPKM・RPKMの場合は使わないほうが良いでしょう。発現差解析をする際には、通常のFold値やP値を用いる方法だけでなく、グループAでは値が無いけどグループBでは値がある遺伝子群、あるいはその逆の遺伝子群も加える必要があります。この分手間がかかりますので、忘れないようにしてください。
ステップ4の説明も上記Centeringと同じですが、注意点は、Controlサンプルで測定値が欠損値となっている可能性があるので、Ratio to Control Samplesブロックへの置き換えはしないほうが良いということです。
ただ、この状態でクラスタリングなどの多変量解析を行うと、欠損値が邪魔になります。そこで、Centeringの後にFill Missing Valuesを追加して、-1など発現減少を表す何らかの値に置き換えるのはありでしょう。ということは、発現差解析のときと多変量解析のときで、Fill Missing Valuesの有無を切り替えることが必要となりますので、これも手間がかかります。