この記事は、FASTQ to VCF ツールを使うための準備のためのものです。
これを始める前に
これを始める前に、必ずRNA-Seq FASTQファイル処理パイプラインの設定のやり方(Windows 版)を完了してください。fastp と HISAT2 は同じなので、ここでは説明を省きます。ここでは、追加の GATK4 のインストールについて説明します。
環境設定パネル
設定のやり方は、ほとんどRNA-Seq FASTQファイル処理パイプラインの設定と同じです。fastp と HISAT2 の実行ファイルをダウンロードして、パスを設定してください。GATK の Resource Bundle はヒトのみの対応となっています (b37/hg19 または Grch38/hg38)。ヒト以外の生物種ではご利用いただけません。
ただし、HISAT2のウェブサイトから提供されているインデックスは染色体名が番号だけになっているので、"chr1"のように染色体名を変更するよう編集したfaファイルから構築し直したものを使わないとGATKの実行中にエラーが起きて止まります。そこで、この問題に対応したGRCh38用の"chr"付き HISAT2 インデックス と GTFファイル を用意しました。zip圧縮されていますので、解凍してお使いください。
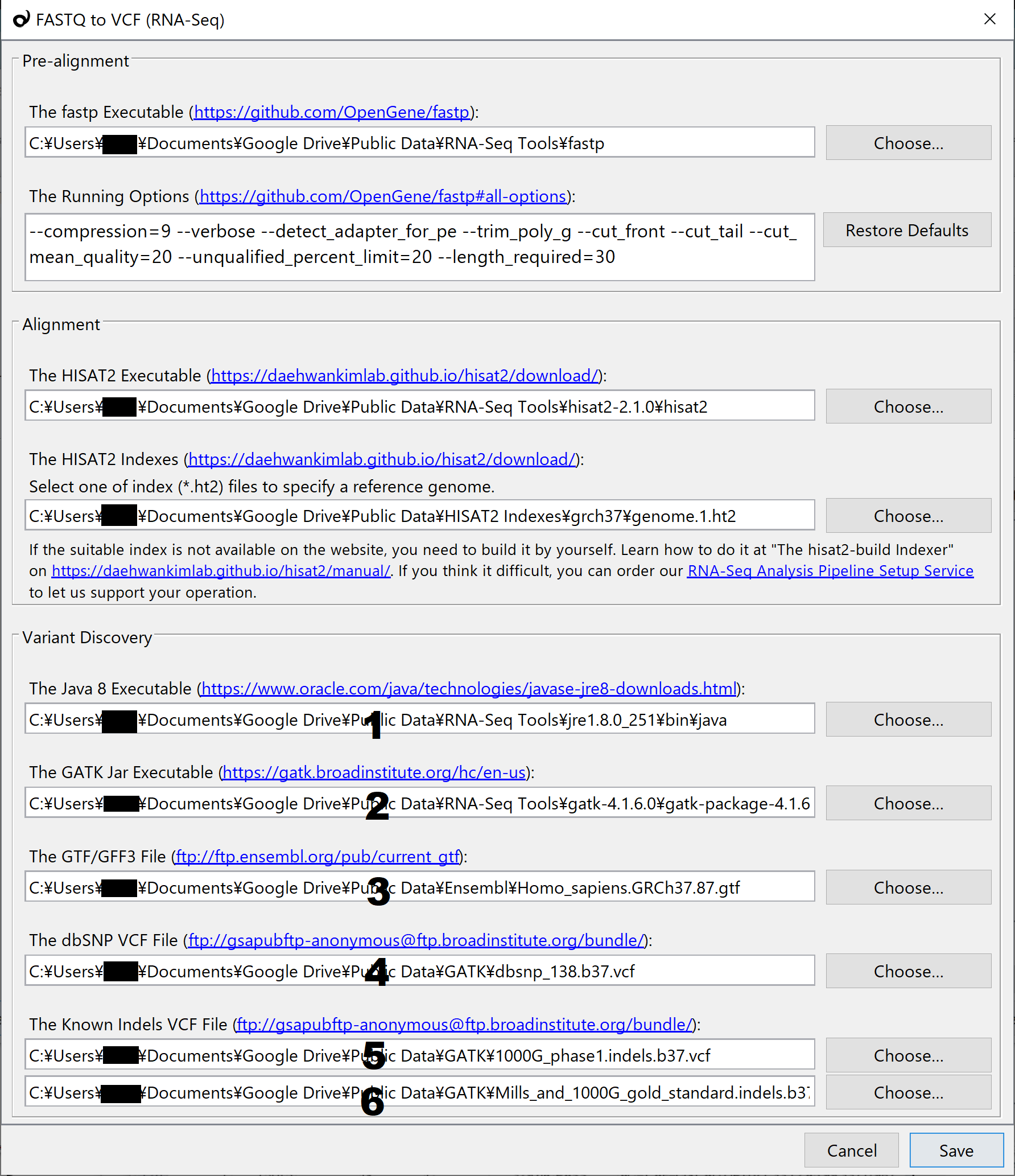
1. Java 8 Executable
Linux x64 Compressed Archive バージョンをダウンロードします。Oracleのアカウントをお持ちでない場合は、サインアップしてください。ダウンロードしたアーカイブファイルを解凍してください。そしたら、設定画面で jdk-[version]\bin\java へのパスを設定してください。
2. GATK jar Executable
最新リリース版の zip ファイルをダウンロードしてください。ダウンロードしたZIPファイルを解凍してください。そしたら、設定画面で gatk[version]\gatk-package-[version]-local.jar へのパスを設定してください。
3. GTF/GFF3 File
grch38版は、こちらからGTFファイルをダウンロードしてください。このGTFファイルは chromosome nameに "chr" を付加している点で、EBIの提供するものとは異なります。ちなみに、このバージョンのGTFで正しく動作させるためには、”chr”付きのHISAT2 Indexとの組合せで使う必要がありますのでご注意ください。
4. dbSNP VCF File
Google Cloud経由で提供されている gcp-public-data--broad-references から取得します。grch38の場合は、hg38/v0 フォルダーの下から、Homo_sapiens_assembly38.dbsnp.vcf をダウンロードしてください。
5&6. Known Indels VCF File
ステップ4と同様に、hg38/v0 フォルダーの下からHomo_sapiens_assembly38.known_indels.vcf と Mills_and_1000G_gold_standard.indels.hg38.vcf をダウンロードしてください。リストにファイル名が見つからない場合は、2ページ目に移動してください。
ステップ4~6のファイルについて注意
Google Cloud上では、".vcf.gz"という拡張子で表示されていますが、ダウンロードしたファイルは ".vcf" などのように変わっていることがあります。生のvcfでもgz圧縮されたファイルでもどちらでも受け付けてくれますが、拡張子を修正しなければいけない場合があります。
hg19で解析する方へ
hg19で解析する場合は、HISAT2 Index (grch37_genome.tar.gz) と GRCh37 GTFファイル はオリジナルのものをお使いください。そして、dbSNPとIndelsファイルは下記のものをお使いください。