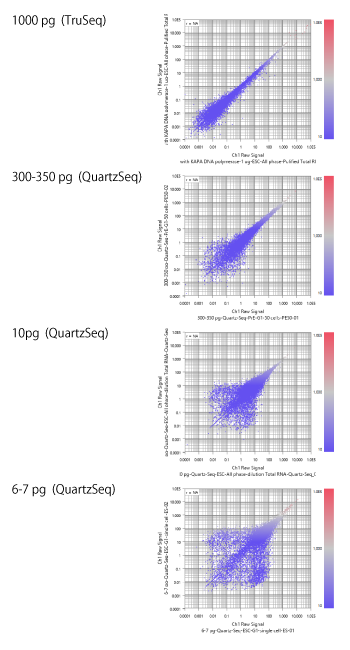
GSE42268は、さまざまな量のインプットRNAが含まれるデータセットです。そこで、インプットの量がRNA-Seqのデータの品質にどのような影響があるかを見てみました。
いちばん上は、1ug の total RNA を測定した結果です。値はFPKMなのでノイズとシグナルの境目がぼやけて見難くはなっていますが、だいたい 0.1 ~ 1 の間に境があるようです。たとえば 0.5 以上を信頼できる測定値として採用するというような判断は妥当のように思います。
上から二つ目は、インプットの量がおよそ 1/3 の 300-350 pg となっています。このデータでは、ノイズとシグナルの境目はおそらく 1 ~ 10 の間にあるでしょう。たとえば 5 以上を信頼できる測定値として採用できそうです。
上から3つ目は、インプットの RNA 量が 10pg となっています。このデータではFPKMの値が 10 より低い領域で一様分布に似てきますので、完全にノイズだと考えられます。値が 50 より下くらいでは点の塊が大きく広がっているので、測定値はまだ信頼できないでしょう。たとえば 50 以上を信頼できる測定値として採用できそうですが、100 以上でないと精度はあまり高くはないようです。
最後はシングルセル、インプットの量が 6 ~ 7pg となっています。このデータではFPKMの値が 100 より低い領域はノイズだと考えられます。精度の高いデータは1000以上のごくわずかな遺伝子だけと言えそうです。
上記をまとめると、次のことが言えるのではないでしょうか。
- インプット RNA の量が少ないほど、ダイナミックレンジは狭く、信頼できる測定値を持つ遺伝子は少なくなる。
- インプット RNA の量やプロトコルなどが違うと FPKM の分布も変わるので、一概に「FPKMが 10 以上なら信頼できる」などの基準を設けられない。