まずはTree Viewを表示したまま操作するとものすごく遅いです。Line Graphはサンプル数が多いと加速度的に遅くなります。従って、できるだけScatter Plot (Merasurements) Viewを表示した状態でさまざまな操作するようにすることで体感スピードが改善します。
大きなデータセットを扱うと、どうしても計算量とディスクアクセスが増えるので遅くなります。ハードウェアでパフォーマンスを改善するには、大容量メモリー、高速なSSD (PCI Express 4.0 x4 NVMe)、高速なCPUがカギとなります。解析に必要なメモリーのサイズについてはこちらをご覧ください。SSDドライブにOSとSubio Platformを入れて動かす場合は、ディスクの空き容量を十分に確保してください。
ハードウェアによる解決が難しい場合は、必ずしも効果があるとは限りませんが下記をお試しください。
他のアプリケーションを閉じる:
たくさんのアプリケーションが起動していると、たとえ走っていなくてもSubio Platformが使えるメモリーが少なくなる可能性があります。できるだけ不要なソフトは終了させるようにしましょう。
Viewとtabを非表示にする:
Turning Views and Tabs On/Off
100万プローブとか、数千サンプルとかあるような巨大なデータセットを扱うと、Subio Platformは非常に遅くなります。
このようなとき、Viewやタブの表示を切ることで、Subio Platformの応答を改善することができます。
解析が終わったら、Viewとタブの表示をもどして見ることができます。
Drawing Mode を Speed にする:
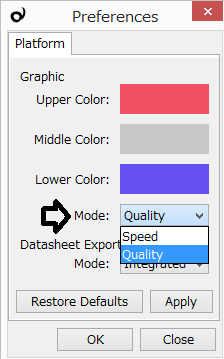
Platform または Plug-in メニューから Preferences 画面を開いて、描画モードをデフォルトの Quality から Speed に変えると、グラフはなめらかでなくなりますが速く描画します。
グラフの軸の最大値と最小値を、実数で設定する:
軸の設定は、デフォルトでは max あるいは min となっていますが、これはその都度表示される最大値と最小値を走査することになります。実数を設定することで、このような処理を省略することができます。