これはMacユーザー向けに、RNA-SeqのFASTQファイルをSubio Platformにインポートするための準備について説明したものです。もしWindowsをお使いでしたらWindows10版の説明をご覧ください。
Subio Platformは、下記のツールを使ってRNA-SeqのFASTQファイルを処理します。
手順:
- Anaconda をダウンロード、およびインストールする。たとえ M1 プロセッサーのコンピューターをお使いの場合でも、明示的に通常版("(M1)" と付記されていない方)のインストーラーをダウンロードしてください。まだ多くのバイオインフォマティクスツールがM1環境に未対応なため、エラーの原因となります。もし、既にM1版のAnacondaをインストールされている場合は、一度それをアンインストールしてください。
- ターミナルを起動し、次のコマンドを入力してfastp、HISAT2、StringTieをインストールする。fastpのインストールコマンドには、必ず 0.22.0のバージョン指定オプションを付けてください。
$ conda install -c bioconda fastp==0.22.0 $ conda install -c bioconda hisat2 $ conda install -c bioconda stringtie
- 下記のコマンドを入力して、fastp、HISAT2、StringTie の実行ファイルのパスを表示する。
$ which fastp $ which hisat2 $ which stringtie
- whichコマンドで表示されたfastp、HISAT2、StringTieの実行ファイルのパスを、Subio Platformの設定画面にコピー&ペーストする。
- 目的の生物種のHISAT2 のインデックス をダウンロードする。
- 目的の生物種の、HISAT2インデックスと同じゲノムバージョンのGTFファイル をダウンロードする。
- Subio Platformの設定画面で、HISAT2のインデックスとGTFファイルのパスを指定する。
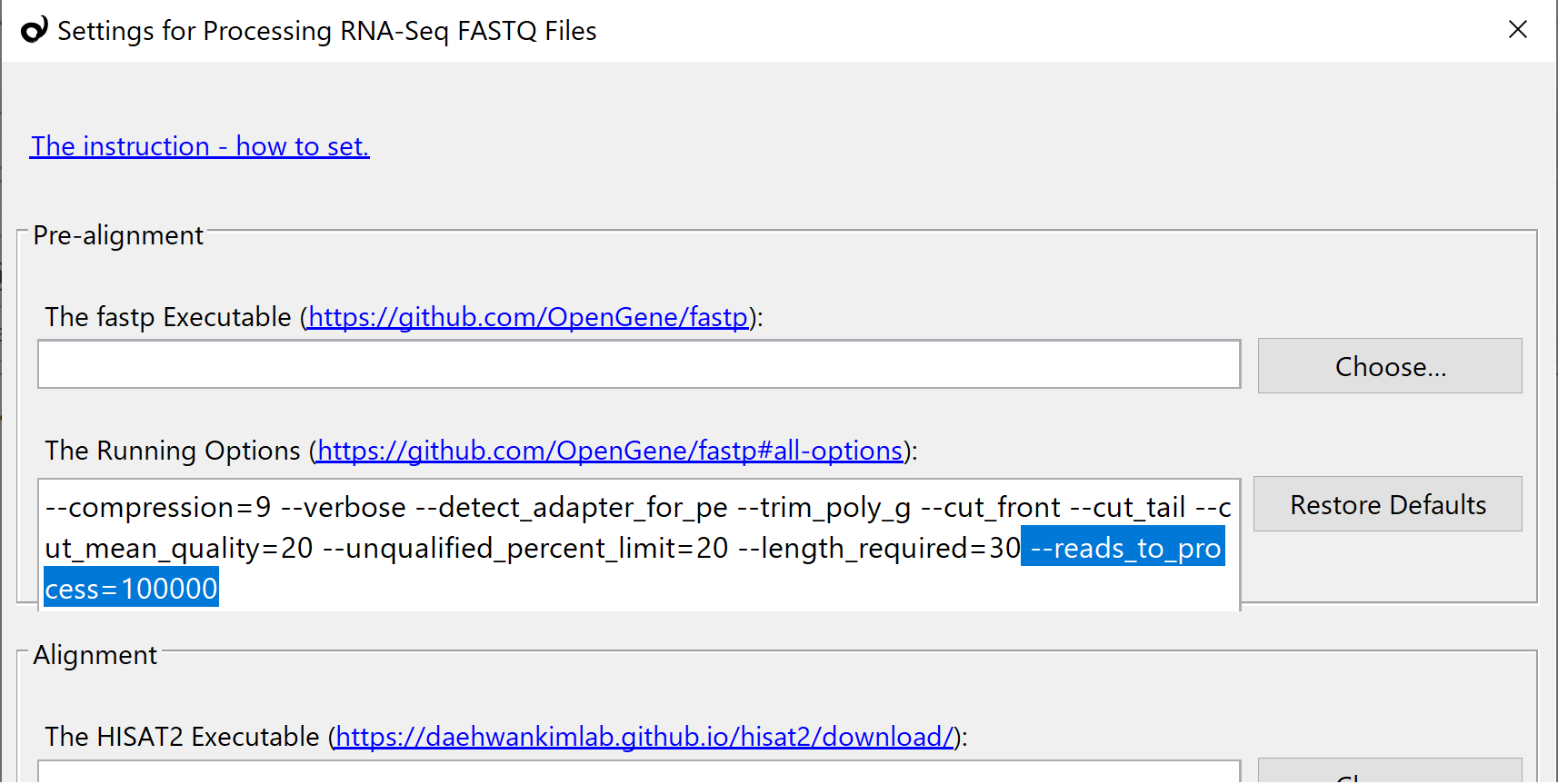
FASTQファイルを処理するのにはとても長い時間がかかります。そこで、パイプラインの設定がうまくいったかどうか短い時間で試すために、下記のオプションをfastpのセクションに追加してください。こうすることで、処理するリードの数を制限することができ、FASTQ処理を実行しても数分で完了します。うまくいくことを確認したら、テストのためにインポートしたサンプルはすべて削除し、このオプションを削除してください。
--reads_to_process=100000
上記で紹介したツールは、それぞれ下記のバージョンで正しく実行できることを確かめています。これと異なるバージョンの場合、エラーが起きる可能性があります。
- fastp 0.22.0
- HISAT2 2.1.0
- StringTie 2.1.1
ちなみに、HISAT2 2.2.0を使う場合はちょっとだけ注意が必要で、indexやGTFのパスからすべてのスペースを除く必要があります。
また、もし0.22.0以外のバージョンのfastpをインストールしてしまって動かないときは、上記のanaconda のインストールコマンドを使って、0.22.0を上書きインストールしてください。